Numpy目前还没有基数排序,所以我想知道是否可以使用现有的numpy函数编写一个基数排序。到目前为止,我已经编写了以下代码,虽然可以工作,但比numpy的快排慢大约10倍。
测试和基准测试:
a = np.random.randint(0, 1e8, 1e6)
assert(np.all(radix_sort(a) == np.sort(a)))
%timeit np.sort(a)
%timeit radix_sort(a)
mask_b循环可以部分向量化,从&中广播出遮罩,并使用带有axis参数的cumsum,但这最终会导致性能下降,可能是由于内存占用增加所致。如果有人能够改进我所做的工作,即使它仍然比
np.sort慢...我也会感兴趣,这更多的是出于智力好奇和对numpy技巧的兴趣。请注意,您可以轻松实现快速计数排序,尽管这仅适用于小整数数据。 编辑1:将
np.arange(n)从循环中取出可以稍微提高性能,但这并不是很令人兴奋。
编辑2:cumsum实际上是多余的(哎呀!),但这个更简单的版本只能在性能方面略微提高。def radix_sort(a):
bit_len = np.max(a).bit_length()
n = len(a)
cached_arange = arange(n)
idx = np.empty(n, dtype=int) # fully overwritten each iteration
for mask_b in xrange(bit_len):
is_one = (a & 2**mask_b).astype(bool)
n_ones = np.sum(is_one)
n_zeros = n-n_ones
idx[~is_one] = cached_arange[:n_zeros]
idx[is_one] = cached_arange[:n_ones] + n_zeros
# next three lines just do: a[idx] = a, but correctly
new_a = np.empty(n, dtype=a.dtype)
new_a[idx] = a
a = new_a
return a
编辑 3:与其循环单个位,如果您在多个步骤中构建 idx,则可以一次循环两个或更多位。使用 2 位有所帮助,我尚未尝试更多:
idx[is_zero] = np.arange(n_zeros)
idx[is_one] = np.arange(n_ones)
idx[is_two] = np.arange(n_twos)
idx[is_three] = np.arange(n_threes)
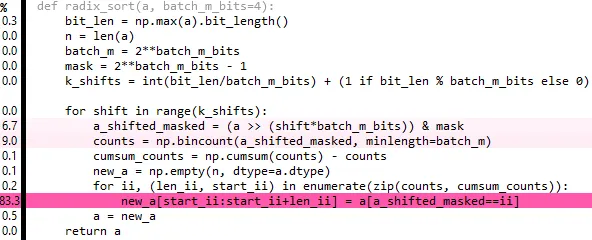
第四次和第五次修改:对于我正在测试的输入来说,将位数设置为4位似乎是最好的选择。此外,您可以完全摆脱idx步骤。现在,与np.sort相比,速度只慢了约5倍,而不是10倍(源代码可在gist中获取):
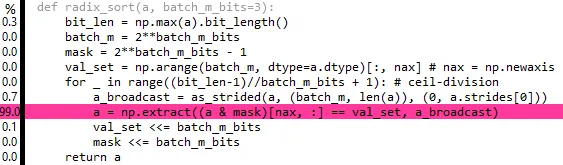
编辑 6: 这是上面内容的整理版,但速度稍微有点慢。80% 的时间都花在了 repeat 和 extract 上 - 如果有一种方式可以广播 extract 就好了 :( ...
def radix_sort(a, batch_m_bits=3):
bit_len = np.max(a).bit_length()
batch_m = 2**batch_m_bits
mask = 2**batch_m_bits - 1
val_set = np.arange(batch_m, dtype=a.dtype)[:, nax] # nax = np.newaxis
for _ in range((bit_len-1)//batch_m_bits + 1): # ceil-division
a = np.extract((a & mask)[nax, :] == val_set,
np.repeat(a[nax, :], batch_m, axis=0))
val_set <<= batch_m_bits
mask <<= batch_m_bits
return a
numpy.lib.stride_tricks中的as_strided广播提取,但在性能方面似乎没有太大帮助:
一开始我觉得这样做是有道理的,因为extract将会迭代整个数组batch_m次,所以CPU请求的缓存行总数与之前相同(只是在整个过程结束时,每个缓存行都被请求了batch_m次)。然而,实际情况是extract无法聪明地迭代任意步长的数组,必须在开始之前扩展数组,即重复操作仍然会发生。事实上,经过查看extract的源代码,我现在看到我们可以采取的最佳方法是:
a = a[np.flatnonzero((a & mask)[nax, :] == val_set) % len(a)]
这比
extract慢一些。但是,如果len(a)是2的幂,我们可以用& (len(a) - 1)替换昂贵的mod操作,这会比extract版本快一些(现在关于a=randint(0, 1e8, 2**20)大约是np.sort的4.9倍)。我想我们可以通过零填充来使非2次幂长度工作,然后在排序结束时裁剪多余的零...但是,除非长度已经接近2的幂,否则这将是一个悲观的选择。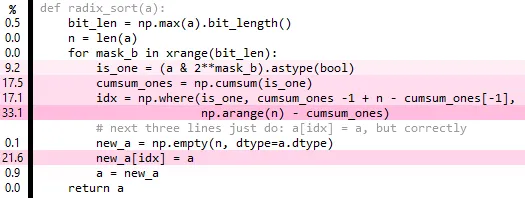
CodeReview,因为在Stackoverflow上有更多的numpy专家。但是对于编码风格和纯 Python 问题,CR 是不错的选择。 - hpauljscipy.sparse.coo.coo_tocsr在 C 语言的基数排序中做了你需要的确切循环... 在适当的条件下,可能比np.sort更快。 - user2379410