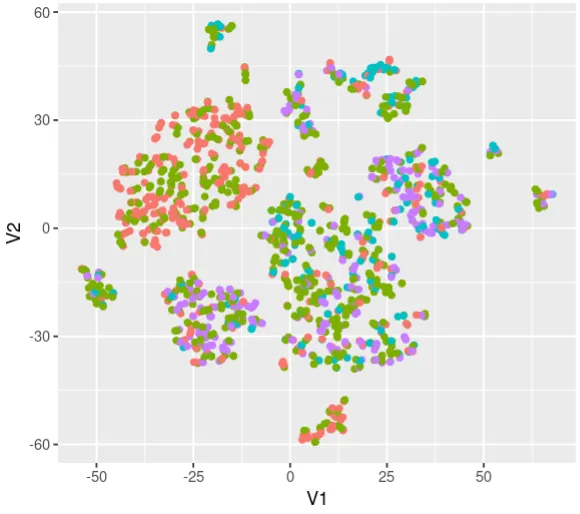
以下图表显示了t-SNE图。我可以在这里展示它,但不幸的是,我无法向您展示标签。有4个不同的标签:
该图使用名为
scores的数据框创建,其中包含约1100个患者样本和25个特征,由其列表示。图的标签来自一个名为metadata的单独数据框。以下代码用于生成图,利用了scores和metadata数据框中的信息。tsneres <- Rtsne(scores, dims = 2, perplexity = 6)
tsneres$Y = as.data.frame(tsneres$Y)
ggplot(tsneres$Y, aes(x = V1, y = V2, color = metadata$labels)) +
geom_point()
我的任务:
我想要分析t-SNE图并确定哪些特征或“scores”矩阵中的列在每个聚类中最为普遍。具体来说,我想要了解哪些特征对于区分图中不同聚类最有帮助。是否可以使用另一种算法,例如保留数据点之间距离的PCA算法来完成此任务?也许这比t-SNE更好?
这是一个示例,这不是真实数据,但它很相似:
structure(list(Feature1 = c(0.1, 0.3, -0.2, -0.12, 0.17, -0.4,
-0.21, -0.19, -0.69, 0.69), Feature2 = c(0.22, 0.42, 0.1, -0.83,
0.75, -0.34, -0.25, -0.78, -0.68, 0.55), Feature3 = c(0.73, -0.2,
0.8, -0.48, 0.56, -0.21, -0.26, -0.78, -0.67, 0.4), Feature4 = c(0.34,
0.5, 0.9, -0.27, 0.64, -0.11, -0.41, -0.82, -0.4, -0.23), Feature5 = c(0.45,
0.33, 0.9, 0.73, 0.65, -0.1, -0.28, -0.78, -0.633, 0.32)), class = "data.frame", row.names = c("Patient_A",
"Patient_B", "Patient_C", "Patient_D", "Patient_E", "Patient_F",
"Patient_G", "Patient_H", "Patient_I", "Patient_J"))
编辑 - PYTHON
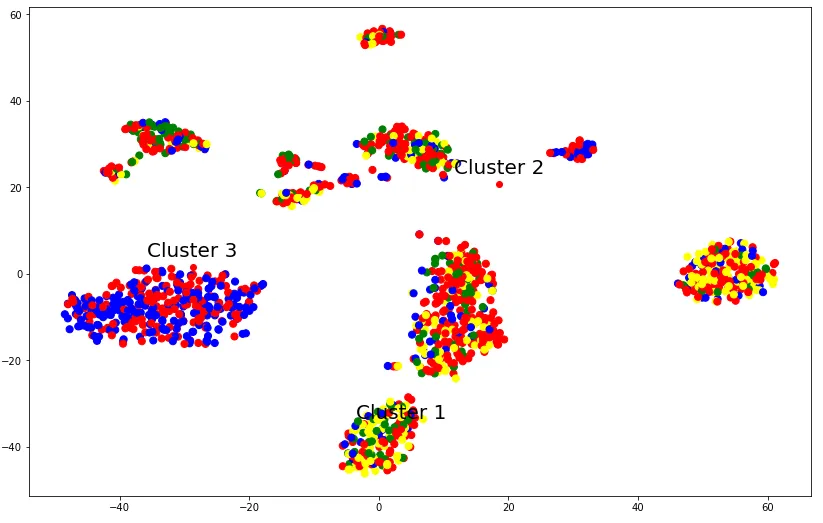
我在Python中达到了相同的程度。我最初尝试了PCA,但它产生了非常糟糕的图形。因此,我首先使用t-SNE降低了维度,这产生了更好的结果,并使用k-means聚类数据。我仍然像以前一样有同样的问题,只是现在我不介意使用R或Python。
这是新图:
这是代码:
from sklearn.manifold import TSNE
tsne = TSNE(n_components=2, perplexity=30, learning_rate=200)
tsne_result = tsne.fit_transform(scores)
#create a dict to map the labels to colors
label_color_dict = {'label1':'blue', 'label2':'red', 'label3':'yellow', 'label4':'green'}
#create a list of colors based on the 'labels' column in metadata
colors = [label_color_dict[label] for label in metadata[['labels']]
plt.scatter(tsne_result[:, 0], tsne_result[:, 1], c=colors, s=50)
plt.scatter(cluster_centers[:, 0], cluster_centers[:, 1], c='red', marker='o')
# Add labels to the cluster centers
for i, center in enumerate(cluster_centers,1):
plt.annotate(f"Cluster {i}", (center[0], center[1]),
textcoords="offset points",
xytext=(0,10), ha='center', fontsize=20)