我写了一个脚本,我认为它应该在Python和R中产生相同的结果,但它们产生了非常不同的答案。每个都试图使用Nelder-Mead最小化偏差来拟合模拟数据。总的来说,R中的优化表现要好得多。我做错了什么吗?R和SciPy中实现的算法不同吗?
Python的结果:
Python的结果:
>>> res = minimize(choiceProbDev, sparams, (stim, dflt, dat, N), method='Nelder-Mead')
final_simplex: (array([[-0.21483287, -1. , -0.4645897 , -4.65108495],
[-0.21483909, -1. , -0.4645915 , -4.65114839],
[-0.21485426, -1. , -0.46457789, -4.65107337],
[-0.21483727, -1. , -0.46459331, -4.65115965],
[-0.21484398, -1. , -0.46457725, -4.65099805]]), array([107.46037865, 107.46037868, 107.4603787 , 107.46037875,
107.46037875]))
fun: 107.4603786452194
message: 'Optimization terminated successfully.'
nfev: 349
nit: 197
status: 0
success: True
x: array([-0.21483287, -1. , -0.4645897 , -4.65108495])
R结果:
> res <- optim(sparams, choiceProbDev, stim=stim, dflt=dflt, dat=dat, N=N,
method="Nelder-Mead")
$par
[1] 0.2641022 1.0000000 0.2086496 3.6688737
$value
[1] 110.4249
$counts
function gradient
329 NA
$convergence
[1] 0
$message
NULL
我已检查过我的代码,据我所知,这似乎是由于优化和最小化之间的某些差异导致的,因为我试图最小化的函数(即choiceProbDev)在每个函数中的操作方式都相同(除了输出外,我还检查了函数中每个步骤的等价性)。例如:
Python的choiceProbDev函数:
>>> choiceProbDev(np.array([0.5, 0.5, 0.5, 3]), stim, dflt, dat, N)
143.31438613033876
R选择概率开发:
> choiceProbDev(c(0.5, 0.5, 0.5, 3), stim, dflt, dat, N)
[1] 143.3144
我也尝试了调整每个优化函数的公差水平,但我不确定这两个参数如何匹配。无论如何,目前我的摆弄并没有使它们达成一致。以下是每个代码的完整内容。
Python:
# load modules
import math
import numpy as np
from scipy.optimize import minimize
from scipy.stats import binom
# initialize values
dflt = 0.5
N = 1
# set the known parameter values for generating data
b = 0.1
w1 = 0.75
w2 = 0.25
t = 7
theta = [b, w1, w2, t]
# generate stimuli
stim = np.array(np.meshgrid(np.arange(0, 1.1, 0.1),
np.arange(0, 1.1, 0.1))).T.reshape(-1,2)
# starting values
sparams = [-0.5, -0.5, -0.5, 4]
# generate probability of accepting proposal
def choiceProb(stim, dflt, theta):
utilProp = theta[0] + theta[1]*stim[:,0] + theta[2]*stim[:,1] # proposal utility
utilDflt = theta[1]*dflt + theta[2]*dflt # default utility
choiceProb = 1/(1 + np.exp(-1*theta[3]*(utilProp - utilDflt))) # probability of choosing proposal
return choiceProb
# calculate deviance
def choiceProbDev(theta, stim, dflt, dat, N):
# restrict b, w1, w2 weights to between -1 and 1
if any([x > 1 or x < -1 for x in theta[:-1]]):
return 10000
# initialize
nDat = dat.shape[0]
dev = np.array([np.nan]*nDat)
# for each trial, calculate deviance
p = choiceProb(stim, dflt, theta)
lk = binom.pmf(dat, N, p)
for i in range(nDat):
if math.isclose(lk[i], 0):
dev[i] = 10000
else:
dev[i] = -2*np.log(lk[i])
return np.sum(dev)
# simulate data
probs = choiceProb(stim, dflt, theta)
# randomly generated data based on the calculated probabilities
# dat = np.random.binomial(1, probs, probs.shape[0])
dat = np.array([0, 0, 0, 0, 0, 0, 1, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 1, 1, 0, 1,
0, 1, 1, 0, 0, 1, 0, 1, 0, 0, 1, 0, 1, 1, 1, 1, 1, 1, 1, 0, 0, 1,
0, 0, 1, 0, 1, 0, 1, 0, 1, 0, 0, 0, 0, 1, 1, 1, 1, 0, 1, 1, 1, 1,
0, 1, 1, 1, 1, 0, 0, 1, 1, 1, 1, 1, 1, 1, 1, 0, 1, 1, 1, 1, 1, 1,
0, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1,
1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1])
# fit model
res = minimize(choiceProbDev, sparams, (stim, dflt, dat, N), method='Nelder-Mead')
R:
library(tidyverse)
# initialize values
dflt <- 0.5
N <- 1
# set the known parameter values for generating data
b <- 0.1
w1 <- 0.75
w2 <- 0.25
t <- 7
theta <- c(b, w1, w2, t)
# generate stimuli
stim <- expand.grid(seq(0, 1, 0.1),
seq(0, 1, 0.1)) %>%
dplyr::arrange(Var1, Var2)
# starting values
sparams <- c(-0.5, -0.5, -0.5, 4)
# generate probability of accepting proposal
choiceProb <- function(stim, dflt, theta){
utilProp <- theta[1] + theta[2]*stim[,1] + theta[3]*stim[,2] # proposal utility
utilDflt <- theta[2]*dflt + theta[3]*dflt # default utility
choiceProb <- 1/(1 + exp(-1*theta[4]*(utilProp - utilDflt))) # probability of choosing proposal
return(choiceProb)
}
# calculate deviance
choiceProbDev <- function(theta, stim, dflt, dat, N){
# restrict b, w1, w2 weights to between -1 and 1
if (any(theta[1:3] > 1 | theta[1:3] < -1)){
return(10000)
}
# initialize
nDat <- length(dat)
dev <- rep(NA, nDat)
# for each trial, calculate deviance
p <- choiceProb(stim, dflt, theta)
lk <- dbinom(dat, N, p)
for (i in 1:nDat){
if (dplyr::near(lk[i], 0)){
dev[i] <- 10000
} else {
dev[i] <- -2*log(lk[i])
}
}
return(sum(dev))
}
# simulate data
probs <- choiceProb(stim, dflt, theta)
# same data as in python script
dat <- c(0, 0, 0, 0, 0, 0, 1, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 1, 1, 0, 1,
0, 1, 1, 0, 0, 1, 0, 1, 0, 0, 1, 0, 1, 1, 1, 1, 1, 1, 1, 0, 0, 1,
0, 0, 1, 0, 1, 0, 1, 0, 1, 0, 0, 0, 0, 1, 1, 1, 1, 0, 1, 1, 1, 1,
0, 1, 1, 1, 1, 0, 0, 1, 1, 1, 1, 1, 1, 1, 1, 0, 1, 1, 1, 1, 1, 1,
0, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1,
1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1)
# fit model
res <- optim(sparams, choiceProbDev, stim=stim, dflt=dflt, dat=dat, N=N,
method="Nelder-Mead")
更新:
在每个迭代中打印估计值后,我现在认为差异可能源于每个算法所采取的“步长”不同。Scipy似乎比optim采取更小的步长(并且初始方向也不同)。我还没有想出如何调整这一点。
Python:
>>> res = minimize(choiceProbDev, sparams, (stim, dflt, dat, N), method='Nelder-Mead')
[-0.5 -0.5 -0.5 4. ]
[-0.525 -0.5 -0.5 4. ]
[-0.5 -0.525 -0.5 4. ]
[-0.5 -0.5 -0.525 4. ]
[-0.5 -0.5 -0.5 4.2]
[-0.5125 -0.5125 -0.5125 3.8 ]
...
R:
> res <- optim(sparams, choiceProbDev, stim=stim, dflt=dflt, dat=dat, N=N, method="Nelder-Mead")
[1] -0.5 -0.5 -0.5 4.0
[1] -0.1 -0.5 -0.5 4.0
[1] -0.5 -0.1 -0.5 4.0
[1] -0.5 -0.5 -0.1 4.0
[1] -0.5 -0.5 -0.5 4.4
[1] -0.3 -0.3 -0.3 3.6
...
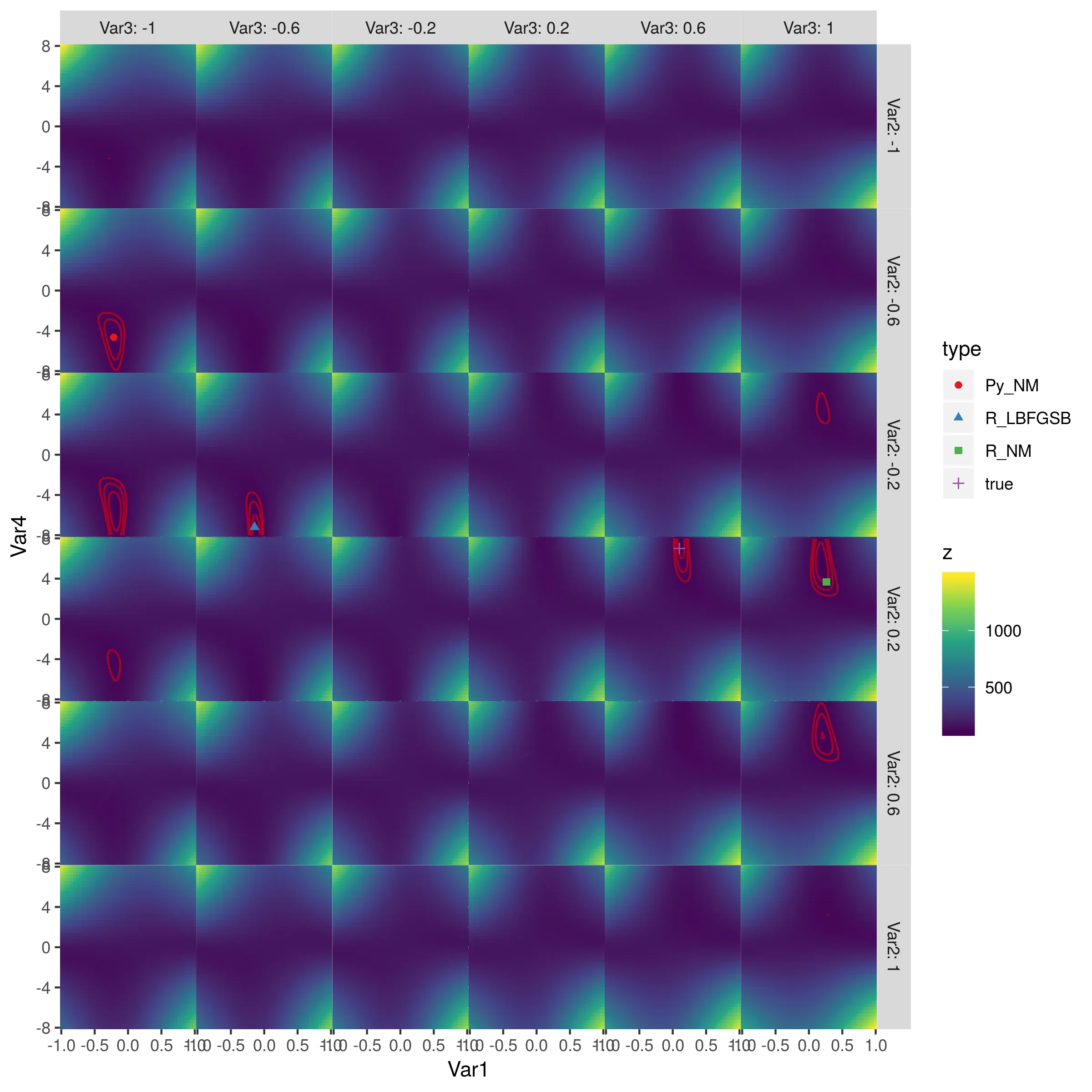
controllist中玩弄args,以与Python的minimize(method='Nelder-Mead')默认值匹配。我认为这是由于默认值不同造成的。 - Parfaitoptim中的 Nelder-Mead 方法并不是 R 中可用的最佳或最准确的实现。您可以尝试使用 dfoptim 包中的nmk[b]或 adagio 中的neldermead[b],或者在 pracma 中尝试自适应版本anms等其他方法。这些实现并没有太大的区别,但在精度和效率方面可能会有显著差异,特别是如果存在多个最小值时。 - Hans W.