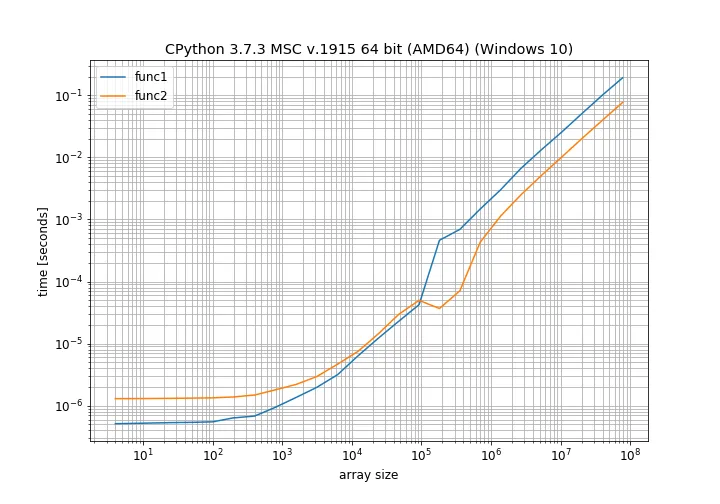
我正在比较numpy数组的原地操作与常规操作。 这是我所做的(使用Python版本3.7.3):
a1, a2 = np.random.random((10,10)), np.random.random((10,10))
def func1(a1, a2):
a1 = a1 + a2
def func2(a1, a2):
a1 += a2
%timeit func1(a1, a2)
%timeit func2(a1, a2)
func1 比 func2 更慢。 但是实际结果是:In [10]: %timeit func1(a1, a2)
595 ns ± 14.4 ns per loop (mean ± std. dev. of 7 runs, 1000000 loops each)
In [11]: %timeit func2(a1, a2)
1.38 µs ± 7.87 ns per loop (mean ± std. dev. of 7 runs, 1000000 loops each)
In [12]: np.__version__
Out[12]: '1.16.2'
这表明func1所需的时间仅为func2所需时间的一半。有人可以帮忙解释一下为什么会这样吗?