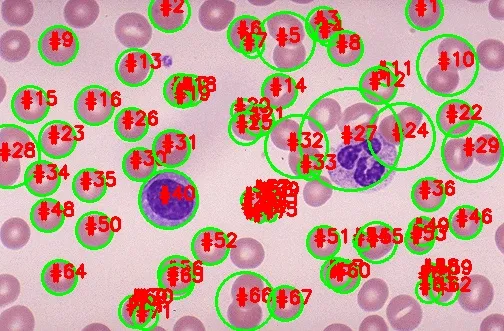
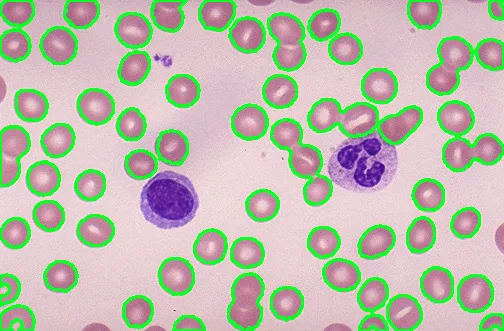
我需要计算图像中粉色细胞的数量的代码,使用阈值分割和分水岭方法。请仅计算粉色的细胞。图片链接如下: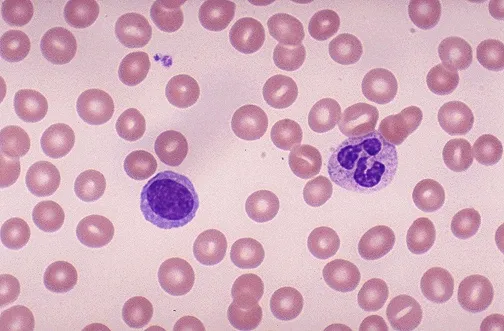

import cv2
from skimage.feature import peak_local_max
from skimage.morphology import watershed
from scipy import ndimage
import numpy as np
import imutils
image = cv2.imread("cellorigin.jpg")
gray = cv2.cvtColor(image, cv2.COLOR_BGR2GRAY)
thresh = cv2.threshold(gray, 0, 255,
cv2.THRESH_BINARY_INV | cv2.THRESH_OTSU)[1]
cv2.imshow("Thresh", thresh)
D = ndimage.distance_transform_edt(thresh)
localMax = peak_local_max(D, indices=False, min_distance=20,
labels=thresh)
cv2.imshow("D image", D)
markers = ndimage.label(localMax, structure=np.ones((3, 3)))[0]
labels = watershed(-D, markers, mask=thresh)
print("[INFO] {} unique segments found".format(len(np.unique(labels)) - 1))
for label in np.unique(labels):
# if the label is zero, we are examining the 'background'
# so simply ignore it
if label == 0:
continue
# otherwise, allocate memory for the label region and draw
# it on the mask
mask = np.zeros(gray.shape, dtype="uint8")
mask[labels == label] = 255
# detect contours in the mask and grab the largest one
cnts = cv2.findContours(mask.copy(), cv2.RETR_EXTERNAL,
cv2.CHAIN_APPROX_SIMPLE)
cnts = imutils.grab_contours(cnts)
c = max(cnts, key=cv2.contourArea)
# draw a circle enclosing the object
((x, y), r) = cv2.minEnclosingCircle(c)
cv2.circle(image, (int(x), int(y)), int(r), (0, 255, 0), 2)
cv2.putText(image, "#{}".format(label), (int(x) - 10, int(y)),
cv2.FONT_HERSHEY_SIMPLEX, 0.6, (0, 0, 255), 2)
cv2.imshow("input",image
cv2.waitKey(0)
我无法正确地将粉色细胞分割开来。在某些地方,两个粉色细胞粘在一起,它们也应该被分开。
输出: