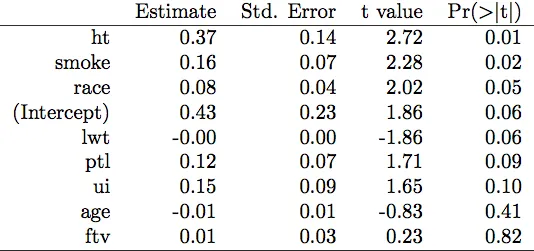
我正在为不同的公司建模大量数据,对于每个公司,我需要快速确定最重要的模型参数。我希望看到一个xtable()输出的拟合模型,按p值递增的顺序排序所有系数(即,最重要的参数首先出现)。
x <- data.frame(a=rnorm(100), b=runif(100), c=rnorm(100), e=rnorm(100))
fit <- glm(a ~ ., data=x)
xtable(fit)
我猜我可以通过更改
fit对象的结构来实现这样的事情。但我对结构不太熟悉,无法自信地进行任何更改。有什么建议吗?
str()是您的好朋友。 :-) - chl