如果事先不知道真实标签(就像您的情况一样),则可以使用拐点准则或轮廓系数来评估 K-Means 聚类。
拐点准则方法:
拐点法背后的思想是在给定数据集上运行 k-means 聚类,对于一系列 k 值(例如 k=1 到 10),为每个 k 值计算平方误差和(SSE)。
然后,绘制每个 k 值的 SSE 的线图。如果线图呈现出手臂状 - 在下面的线图中表示为红色圆圈(如角度)- 手臂上的"拐点"即为最佳 k(聚类数)的值。
这里,我们希望最小化 SSE。随着增加 k,SSE 倾向于逐渐减少并趋近于 0(当 k 等于数据集中的数据点数时,SSE 为 0,因为每个数据点都是其自身的聚类,与其聚类中心之间没有误差)。
因此,目标是选择一个较小的 k 值,仍具有较低的 SSE,而拐点通常表示通过增加 k 值获得收益递减的位置。
让我们考虑鸢尾花数据集,
import pandas as pd
from sklearn.datasets import load_iris
from sklearn.cluster import KMeans
import matplotlib.pyplot as plt
iris = load_iris()
X = pd.DataFrame(iris.data, columns=iris['feature_names'])
data = X[['sepal length (cm)', 'sepal width (cm)', 'petal length (cm)']]
sse = {}
for k in range(1, 10):
kmeans = KMeans(n_clusters=k, max_iter=1000).fit(data)
data["clusters"] = kmeans.labels_
sse[k] = kmeans.inertia_
plt.figure()
plt.plot(list(sse.keys()), list(sse.values()))
plt.xlabel("Number of cluster")
plt.ylabel("SSE")
plt.show()
上述代码的图表如下:
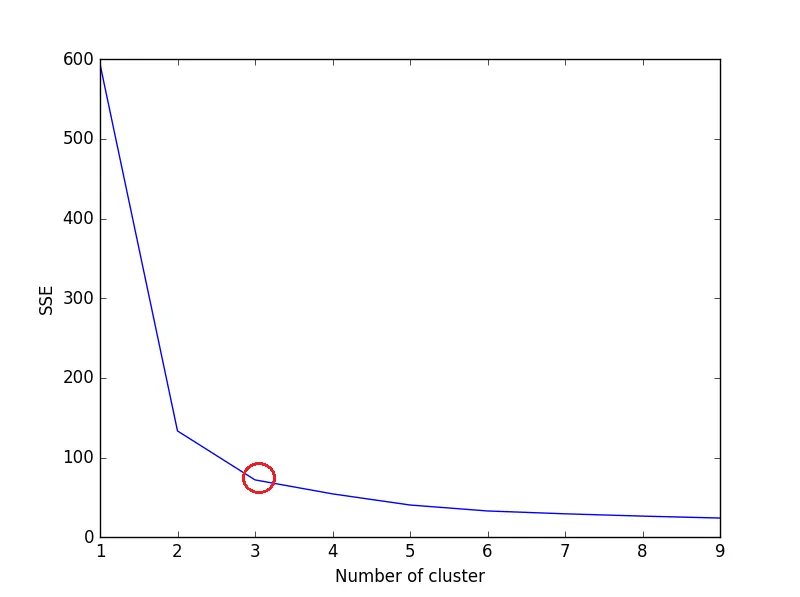
在图表中,我们可以看到对于鸢尾花数据集,“3”是最佳聚类数(用红圈圈出来),这是正确的。
轮廓系数法:
从 sklearn 文档 中可以得知,轮廓系数越高,表示聚类效果越好。轮廓系数被定义为每个样本点的两个分数之差:
`
a:一个样本与同一类中所有其他样本的平均距离。
b:一个样本与其它最近的类中所有样本的平均距离。
单个样本点的轮廓系数定义如下:
})
现在,为了找到适合 KMeans 的最优值 k,需要在 1..n 中循环 n_clusters,并为每个样本计算轮廓系数。
更高的轮廓系数表明对象与其自身聚类匹配良好,但与相邻聚类匹配差。
from sklearn.metrics import silhouette_score
from sklearn.datasets import load_iris
from sklearn.cluster import KMeans
X = load_iris().data
y = load_iris().target
for n_cluster in range(2, 11):
kmeans = KMeans(n_clusters=n_cluster).fit(X)
label = kmeans.labels_
sil_coeff = silhouette_score(X, label, metric='euclidean')
print("For n_clusters={}, The Silhouette Coefficient is {}".format(n_cluster, sil_coeff))
输出 -
对于n_clusters=2,轮廓系数为0.680813620271。
对于n_clusters=3,轮廓系数为0.552591944521。
对于n_clusters=4,轮廓系数为0.496992849949。
对于n_clusters=5,轮廓系数为0.488517550854。
对于n_clusters=6,轮廓系数为0.370380309351。
对于n_clusters=7,轮廓系数为0.356303270516。
对于n_clusters=8,轮廓系数为0.365164535737。
对于n_clusters=9,轮廓系数为0.346583642095。
对于n_clusters=10,轮廓系数为0.328266088778。
从这些结果可以看出,n_clusters=2具有最高的轮廓系数。这意味着2应该是最优数量的簇点,对吗?
但问题在于:
Iris数据集有3种花的品种,这与将2作为最优簇点数相矛盾。因此,尽管n_clusters=2具有最高的轮廓系数,由于以下原因,我们会考虑n_clusters=3作为最优簇点数:
- Iris数据集有3种品种。(最重要的)
- n_clusters=3具有第二高的轮廓系数。
因此,选择n_clusters=3是Iris数据集的最佳聚类数量。
选择最优聚类数量将取决于数据集的类型和我们试图解决的问题。但在大多数情况下,选择最高的轮廓系数将产生最优簇点数。
希望这有所帮助!

轮廓分数,但出现了这个错误:ValueError: Number of labels is 1. Valid values are 2 to n_samples - 1 (inclusive)。有什么想法,可能是什么原因呢?我也已经在这个问题上提出了一个问题https://dev59.com/rlUK5IYBdhLWcg3wuB54。 - Suhail Gupta