我有一个数据框,其中一列包含长度不同的列表:
IP <- structure(list(V1 = list(l1 = c("M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M"), `l2` = c("D",
"D", "D", "D", "D", "D", "D", "D", "D", "D", "D", "D", "D", "D",
"D", "D", "D", "D", "D", "D", "D", "D", "D", "D", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M"), `l3` = c("D",
"D", "D", "D", "D", "D", "D", "D", "D", "D", "D", "D", "D", "D",
"D", "D", "D", "D", "D", "D", "D", "D", "D", "D", "D", "D", "D",
"D", "D", "D", "D", "D", "D", "D", "D", "D", "D", "D", "D", "D",
"D", "D", "D", "D", "D", "D", "D", "D", "D", "D", "D", "D", "D",
"D", "D", "D", "D", "D", "D", "D", "D", "D", "D", "D", "D", "D",
"D", "D", "D", "D", "D", "D", "D", "D", "D", "D", "D", "D", "D",
"D", "D", "D", "D", "D", "D", "D", "D", "D", "D", "D", "D", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M", "M",
"M"))), class = "data.frame", row.names = c("1", "2", "3"))
我正在使用以下命令来转换列表。对于较小的数据集,这个命令可以正常工作。
output <- plyr::ldply(IP$V1, rbind)
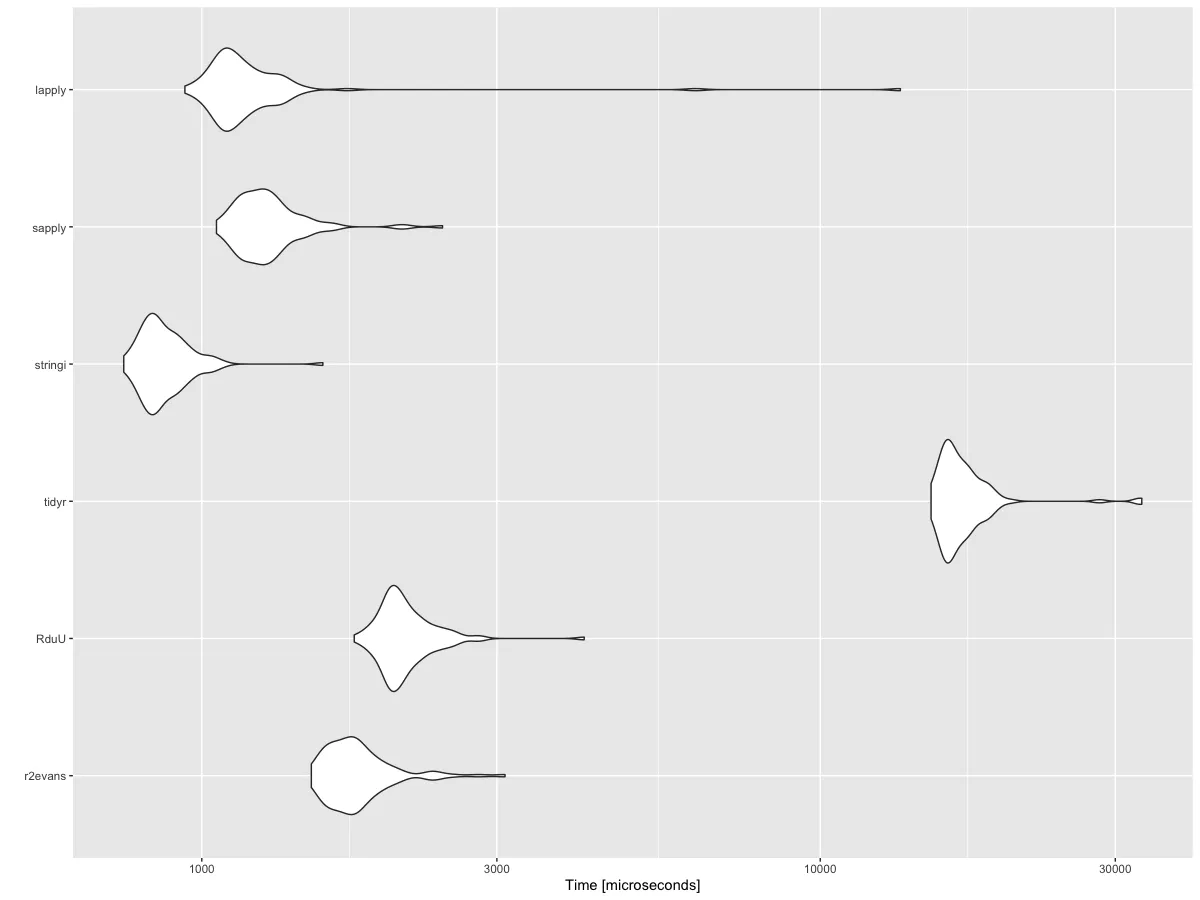
但是,当我将这个应用到一个大数据集(大约>100万)时,它会运行很长时间并崩溃。
有没有一种更有效的方法来处理更大的数据集?
谢谢!