我希望找到一种方法,可以将不同级别的分别计算的lm(a~b)函数的残差添加到我的数据表中作为一列。
有人建议我使用sort_by(c)函数,但这似乎无法与lm(a~b)一起使用。
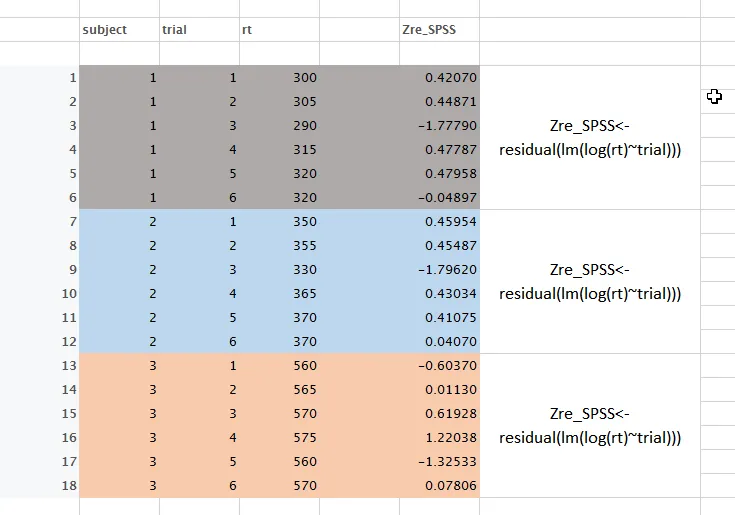
我的工作示例数据如下:
列名为subject、trial和rt的数据框(data.frame)中,我的目标是从一个R函数计算出我最初在SPSS中创建的Zre_SPSS。
我已经尝试过:
data %<>% group_by (subject) %>%
mutate(Zre=residuals(lm(log(rt)~trial)))
但它不起作用- Zre得到计算,但不是分别针对每个主题进行计算,而是针对整个数据框。
请问有人能帮我吗?我完全是R(以及编码)的新手,所以如果这个问题很蠢或者是duplicate,请原谅我,可能是我没有理解其他解决方案,或者它们不是我寻找的解决方案。最好的祝福。
按照Ben Bolker的要求,这里是生成来自Excel屏幕截图数据的R代码
#generate data
subject<-c(1,1,1,1,1,1,2,2,2,2,2,2,3,3,3,3,3,3)
subject<-factor(subject)
trial<-c(1,2,3,4,5,6,1,2,3,4,5,6,1,2,3,4,5,6)
rt<-c(300,305,290,315,320,320,350,355,330,365,370,370,560,565,570,575,560,570)
#Following variable is what I would get after using SPSS code
ZreSPSS<-c(0.4207,0.44871,-1.7779,0.47787,0.47958,-0.04897,0.45954,0.45487,-1.7962,0.43034,0.41075,0.0407,-0.6037,0.0113,0.61928,1.22038,-1.32533,0.07806)
#make data frame
sym<-data.frame(subject, trial, rt, ZreSPSS)
tidyr::nest和一个快速博客简介。 - r2evanstrial而不是作为因子的模型似乎没有太多意义。这真的是您在 SPSS 模型上拟合的数据吗? - Hong Ooiln(log(rt)~trial)预测log(rt)(延迟),并保存对应于每个 rt 的残差。 - blazej