问题: 我如何使用 bootstrap 方法为数据帧中每个组(因子水平)计算的协方差矩阵的特征值上的一系列统计量获取置信区间?
问题描述: 我无法确定我需要的数据结构以便在boot函数中包含这些结果,或者一种“映射”bootstrap方法到各个组,并获得适合绘图的置信区间的方法。
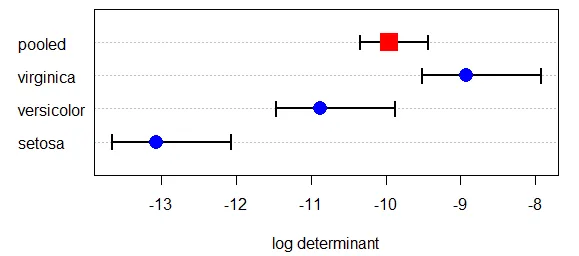
背景说明: 在heplots软件包中,boxM函数计算协方差矩阵相等的 Box's M 测试。该软件包提供了一个绘图函数可以绘制用于该测试的对数行列式的有用图形。该图形中的置信区间基于渐近理论逼近所得。
> library(heplots)
> iris.boxm <- boxM(iris[, 1:4], iris[, "Species"])
> iris.boxm
Box's M-test for Homogeneity of Covariance Matrices
data: iris[, 1:4]
Chi-Sq (approx.) = 140.94, df = 20, p-value < 2.2e-16
> plot(iris.boxm, gplabel="Species")
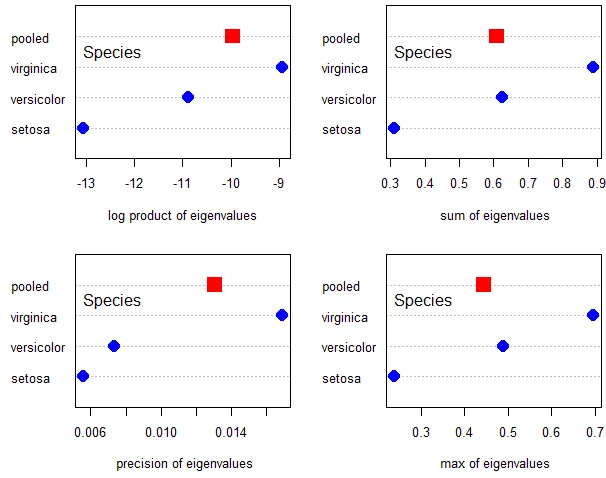
plot方法还可以显示特征值的其他函数,但在这种情况下没有可用的理论置信区间。
op <- par(mfrow=c(2,2), mar=c(5,4,1,1))
plot(iris.boxm, gplabel="Species", which="product")
plot(iris.boxm, gplabel="Species", which="sum")
plot(iris.boxm, gplabel="Species", which="precision")
plot(iris.boxm, gplabel="Species", which="max")
par(op)
因此,我希望能够使用一种引导方法来计算这些置信区间,并在相应的图中显示它们。
我尝试过的方法:
以下是引导这些统计量的函数,但仅针对整个样本,而不考虑组 (Species)。
cov_stat_fun <- function(data, indices,
stats=c("logdet", "prod", "sum", "precision", "max")
) {
dat <- data[indices,]
cov <- cov(dat, use="complete.obs")
eigs <- eigen(cov)$values
res <- c(
"logdet" = log(det(cov)),
"prod" = prod(eigs),
"sum" = sum(eigs),
"precision" = 1/ sum(1/eigs),
"max" = max(eigs)
)
}
boot_cov_stat <- function(data, R=500, ...) {
boot(data, cov_stat_fun, R=R, ...)
}
这个方法是可行的,但我需要按组别(以及总样本)来获取结果。
> iris.boot <- boot_cov_stat(iris[,1:4])
>
> iris.ci <- boot.ci(iris.boot)
> iris.ci
BOOTSTRAP CONFIDENCE INTERVAL CALCULATIONS
Based on 500 bootstrap replicates
CALL :
boot.ci(boot.out = iris.boot)
Intervals :
Level Normal Basic Studentized
95% (-6.622, -5.702 ) (-6.593, -5.653 ) (-6.542, -5.438 )
Level Percentile BCa
95% (-6.865, -5.926 ) (-6.613, -5.678 )
Calculations and Intervals on Original Scale
Some BCa intervals may be unstable
>
我还编写了一个函数,用于计算每个组的单独协方差矩阵,但我不知道如何在我的自助函数中使用它。有人能帮忙吗?
# calculate covariance matrices by group and pooled
covs <- function(Y, group) {
Y <- as.matrix(Y)
gname <- deparse(substitute(group))
if (!is.factor(group)) group <- as.factor(as.character(group))
valid <- complete.cases(Y, group)
if (nrow(Y) > sum(valid))
warning(paste(nrow(Y) - sum(valid)), " cases with missing data have been removed.")
Y <- Y[valid,]
group <- group[valid]
nlev <- nlevels(group)
lev <- levels(group)
mats <- aux <- list()
for(i in 1:nlev) {
mats[[i]] <- cov(Y[group == lev[i], ])
}
names(mats) <- lev
pooled <- cov(Y)
c(mats, "pooled"=pooled)
}
编辑:
在一个似乎相关的问题中,按组引导,建议使用boot()中的strata参数提供答案,但是没有展示它所给出的示例。[啊:仅确保在引导样本中以与数据中其频率相关的方式表示层次结构的strata参数。]
对于我的问题尝试这个,我并没有得到更多启示,因为我想要获取的是每个Species的单独置信区间。
> iris.boot.strat <- boot_cov_stat(iris[,1:4], strata=iris$Species)
>
> boot.ci(iris.boot.strat, conf=0.95, type=c("basic", "bca"))
BOOTSTRAP CONFIDENCE INTERVAL CALCULATIONS
Based on 500 bootstrap replicates
CALL :
boot.ci(boot.out = iris.boot.strat, conf = 0.95, type = c("basic",
"bca"))
Intervals :
Level Basic BCa
95% (-6.587, -5.743 ) (-6.559, -5.841 )
Calculations and Intervals on Original Scale
Some BCa intervals may be unstable
>
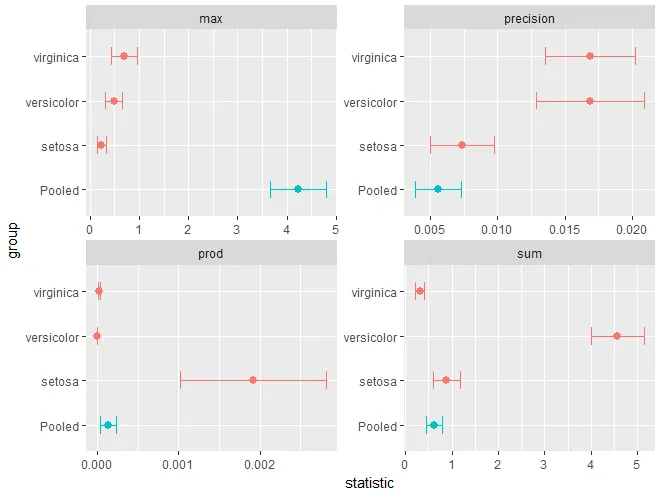
broom::tidy(boot.list)的东西的结果,即作为可用于绘图的数据框的结果。 - user101089