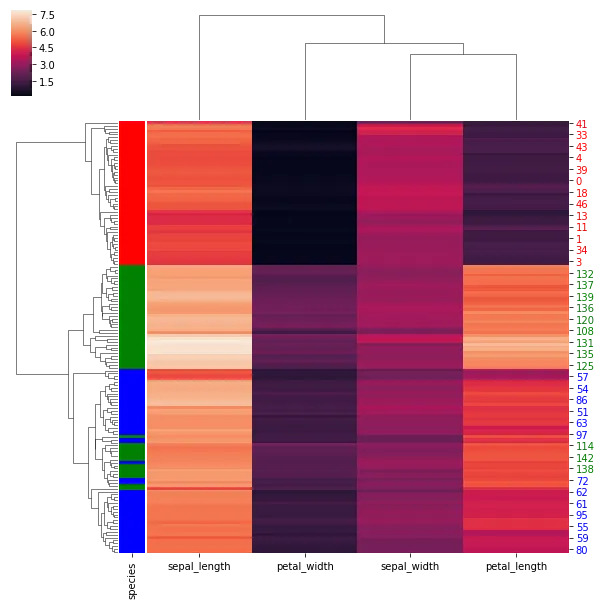
在 seaborn.clustermap 中,是否可以更改 ytick 标签的颜色?
因此,在 seaborn 鸢尾花示例 中,可以根据物种设置行颜色并绘制聚类图:
import seaborn as sns
iris = sns.load_dataset("iris")
species = iris.pop("species")
lut = dict(zip(species.unique(), "rbg"))
row_colors = species.map(lut)
g = sns.clustermap(iris)
可以在绘制的行和行标签之间建立一对一的对应关系:
g.ax_heatmap.yaxis.get_majorticklabels()
有没有办法利用这个功能根据行颜色重新着色ytick标签?