I'll share a couple of commands in the script for examples. In each of the examples, the datasets are 10 to 15 million rows and 10 to 15 columns.
1. 获取由九个变量分组的数据框的最低日期
dataframe %>%
group_by(key_a, key_b, key_c,
key_d, key_e, key_f,
key_g, key_h, key_i) %>%
summarize(min_date = min(date)) %>%
ungroup()
在两个数据帧上执行左连接以添加额外列。
merge(dataframe,
dataframe_two,
by = c("key_a", "key_b", "key_c",
"key_d", "key_e", "key_f",
"key_g", "key_h", "key_i"),
all.x = T) %>%
as_tibble()
dataframe %>%
left_join(dataframe_two,
by = "key_a") %>%
group_by(key_a, date.x) %>%
summarise(key_z = key_z[which.min(abs(date.x - date.y))]) %>%
arrange(date.x) %>%
rename(day = date.x)
我可以应用哪些最佳实践,特别是针对大型数据集,我可以做什么来优化这些类型的函数?
--
这是一个示例数据集。set.seed(1010)
library("conflicted")
conflict_prefer("days", "lubridate")
bigint <- rep(
sample(1238794320934:19082323109, 1*10^7)
)
key_a <-
rep(c("green", "blue", "orange"), 1*10^7/2)
key_b <-
rep(c("yellow", "purple", "red"), 1*10^7/2)
key_c <-
rep(c("hazel", "pink", "lilac"), 1*10^7/2)
key_d <-
rep(c("A", "B", "C"), 1*10^7/2)
key_e <-
rep(c("D", "E", "F", "G", "H", "I"), 1*10^7/5)
key_f <-
rep(c("Z", "M", "Q", "T", "X", "B"), 1*10^7/5)
key_g <-
rep(c("Z", "M", "Q", "T", "X", "B"), 1*10^7/5)
key_h <-
rep(c("tree", "plant", "animal", "forest"), 1*10^7/3)
key_i <-
rep(c("up", "up", "left", "left", "right", "right"), 1*10^7/5)
sequence <-
seq(ymd("2010-01-01"), ymd("2020-01-01"), by = "1 day")
date_sequence <-
rep(sequence, 1*10^7/(length(sequence) - 1))
dataframe <-
data.frame(
bigint,
date = date_sequence[1:(1*10^7)],
key_a = key_a[1:(1*10^7)],
key_b = key_b[1:(1*10^7)],
key_c = key_c[1:(1*10^7)],
key_d = key_d[1:(1*10^7)],
key_e = key_e[1:(1*10^7)],
key_f = key_f[1:(1*10^7)],
key_g = key_g[1:(1*10^7)],
key_h = key_h[1:(1*10^7)],
key_i = key_i[1:(1*10^7)]
)
dataframe_two <-
dataframe %>%
mutate(date_sequence = ymd(date_sequence) + days(1))
sequence_sixdays <-
seq(ymd("2010-01-01"), ymd("2020-01-01"), by = "6 days")
date_sequence <-
rep(sequence_sixdays, 3*10^6/(length(sequence_sixdays) - 1))
key_z <-
sample(1:10000000, 3*10^6)
dataframe_three <-
data.frame(
key_a = sample(key_a, 3*10^6),
date = date_sequence[1:(3*10^6)],
key_z = key_z[1:(3*10^6)]
)
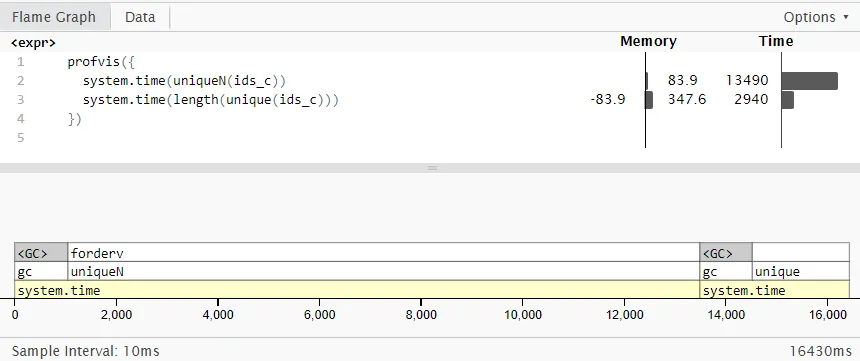
microbenchmark进行测试的脚本吗? - Walditidyft::parse_fst函数,该函数用于读取 fst 文件。 - akrunlubridate包,它使用ymd函数。总体上,这个问题可以通过改进使其完全可复制,这对于回答者提供工作代码是有用的。 - jangorecki