对于那些不熟悉的人来说,独热编码只是将类别列(即因子)转换为多个二进制指示变量列的过程,其中每个新列对应原始列的一个类别。以下示例将更好地说明:
dt <- data.table(
ID=1:5,
Color=factor(c("green", "red", "red", "blue", "green"), levels=c("blue", "green", "red", "purple")),
Shape=factor(c("square", "triangle", "square", "triangle", "cirlce"))
)
dt
ID Color Shape
1: 1 green square
2: 2 red triangle
3: 3 red square
4: 4 blue triangle
5: 5 green cirlce
# one hot encode the colors
color.binarized <- dcast(dt[, list(V1=1, ID, Color)], ID ~ Color, fun=sum, value.var="V1", drop=c(TRUE, FALSE))
# Prepend Color_ in front of each one-hot-encoded feature
setnames(color.binarized, setdiff(colnames(color.binarized), "ID"), paste0("Color_", setdiff(colnames(color.binarized), "ID")))
# one hot encode the shapes
shape.binarized <- dcast(dt[, list(V1=1, ID, Shape)], ID ~ Shape, fun=sum, value.var="V1", drop=c(TRUE, FALSE))
# Prepend Shape_ in front of each one-hot-encoded feature
setnames(shape.binarized, setdiff(colnames(shape.binarized), "ID"), paste0("Shape_", setdiff(colnames(shape.binarized), "ID")))
# Join one-hot tables with original dataset
dt <- dt[color.binarized, on="ID"]
dt <- dt[shape.binarized, on="ID"]
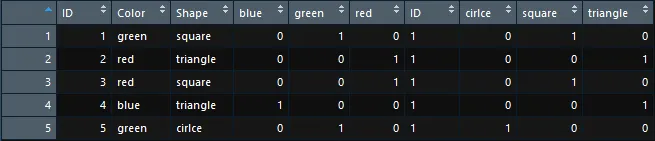
dt
ID Color Shape Color_blue Color_green Color_red Color_purple Shape_cirlce Shape_square Shape_triangle
1: 1 green square 0 1 0 0 0 1 0
2: 2 red triangle 0 0 1 0 0 0 1
3: 3 red square 0 0 1 0 0 1 0
4: 4 blue triangle 1 0 0 0 0 0 1
5: 5 green cirlce 0 1 0 0 1 0 0
这是我经常做的事情,但是你可以看到它非常繁琐(特别是当我的数据有许多因子列时)。 有没有更简单的方法使用data.table完成? 特别是,当我尝试像这样进行一些操作时,我认为dcast将允许我同时对多个列进行独热编码:
这是我经常做的事情,但是你可以看到它非常繁琐(特别是当我的数据有许多因子列时)。 是否有更简便的方式使用data.table完成此操作? 特别是,我假设dcast可以一次性对多个列进行独热编码:
dcast(dt[, list(V1=1, ID, Color, Shape)], ID ~ Color + Shape, fun=sum, value.var="V1", drop=c(TRUE, FALSE))
我获取列组合
ID blue_cirlce blue_square blue_triangle green_cirlce green_square green_triangle red_cirlce red_square red_triangle purple_cirlce purple_square purple_triangle
1: 1 0 0 0 0 1 0 0 0 0 0 0 0
2: 2 0 0 0 0 0 0 0 0 1 0 0 0
3: 3 0 0 0 0 0 0 0 1 0 0 0 0
4: 4 0 0 1 0 0 0 0 0 0 0 0 0
5: 5 0 0 0 1 0 0 0 0 0 0 0 0
dcast(melt(dt, 1), ID ~ value, length)。这个问题可能已经有人解决过了。 - David Arenburg?model.matrix- Hong Ooi