我正在为我的无向网络计算三元组普查,方法如下。
import networkx as nx
G = nx.Graph()
G.add_edges_from(
[('A', 'B'), ('A', 'C'), ('D', 'B'), ('E', 'C'), ('E', 'F'),
('B', 'H'), ('B', 'G'), ('B', 'F'), ('C', 'G')])
from itertools import combinations
#print(len(list(combinations(G.nodes, 3))))
triad_class = {}
for nodes in combinations(G.nodes, 3):
n_edges = G.subgraph(nodes).number_of_edges()
triad_class.setdefault(n_edges, []).append(nodes)
print(triad_class)
它对小型网络的处理效果良好。然而,现在我有一个大一点的网络,约有4000-8000个节点。当我用1000个节点的网络运行我的现有代码时,需要几天的时间才能运行完。是否有更有效率的方法来处理这个问题?
我的当前网络大部分是稀疏的。即节点之间只有少量连接。在这种情况下,我可以先留下未连接的节点进行计算,然后将未连接的节点添加到输出中吗?
我也很乐意获得不必计算每个组合的近似答案。
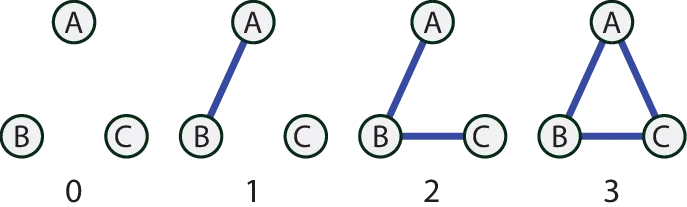
三元组统计的示例:
三元组统计是将三元组(3个节点)分为下图所示的四类。
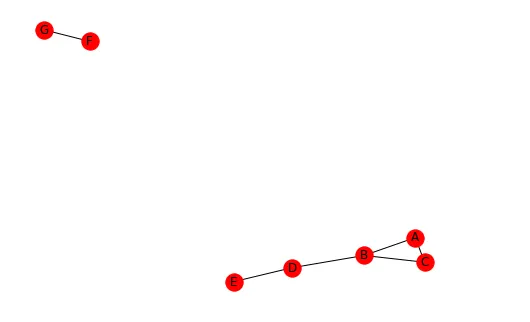
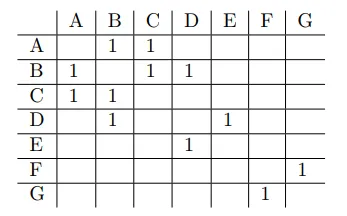
例如考虑下面的网络。
四个类别的三元组统计结果如下:
{3: [('A', 'B', 'C')],
2: [('A', 'B', 'D'), ('B', 'C', 'D'), ('B', 'D', 'E')],
1: [('A', 'B', 'E'), ('A', 'B', 'F'), ('A', 'B', 'G'), ('A', 'C', 'D'), ('A', 'C', 'E'), ('A', 'C', 'F'), ('A', 'C', 'G'), ('A', 'D', 'E'), ('A', 'F', 'G'), ('B', 'C', 'E'), ('B', 'C', 'F'), ('B', 'C', 'G'), ('B', 'D', 'F'), ('B', 'D', 'G'), ('B', 'F', 'G'), ('C', 'D', 'E'), ('C', 'F', 'G'), ('D', 'E', 'F'), ('D', 'E', 'G'), ('D', 'F', 'G'), ('E', 'F', 'G')],
0: [('A', 'D', 'F'), ('A', 'D', 'G'), ('A', 'E', 'F'), ('A', 'E', 'G'), ('B', 'E', 'F'), ('B', 'E', 'G'), ('C', 'D', 'F'), ('C', 'D', 'G'), ('C', 'E', 'F'), ('C', 'E', 'G')]}
如果需要,我很乐意提供更多细节。
编辑:
我按照答案中的建议注释了代码行#print(len(list(combinations(G.nodes, 3)))),成功解决了内存错误(memory error)问题。但即使对于1000个节点的网络,我的程序仍然运行缓慢,需要数天时间。我正在寻找更有效的Python解决方案。
我不限于使用networkx库,也可以接受使用其他库和语言的答案。
如常,如果需要,我很乐意提供更多细节。
g = nx.generators.fast_gnp_random_graph(1000, 0.1, seed=42)。 - EmJ