我正在使用一个
这是一个玩具级别的例子:
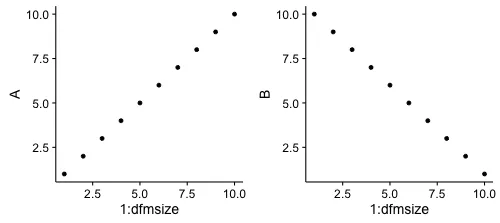
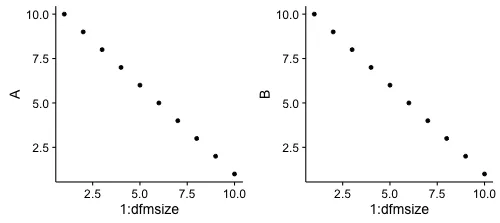
for循环将ggplot分配给列表,然后将其传递给plot_grid()(包cowplot)。plot_grid可以将多个ggplots并排在同一张图中。手动操作没问题,但当我使用for循环时,生成的最后一个图形会在每个子框架中重复显示(如下所示)。换句话说,所有的子框架都显示相同的ggplot。这是一个玩具级别的例子:
require(cowplot)
dfrm <- data.frame(A=1:10, B=10:1)
v <- c("A","B")
dfmsize <- nrow(dfrm)
myplots <- vector("list",2)
count = 1
for(i in v){
myplots[[count]] <- ggplot(dfrm, aes(x=1:dfmsize, y=dfrm[,i])) + geom_point() + labs(y=i)
count = count +1
}
plot_grid(plotlist=myplots)
预期结果:
for loop的图表:
我尝试将列表元素转换为grobs,就像这个问题中描述的那样:
mygrobs <- lapply(myplots, ggplotGrob)
plot_grid(plotlist=mygrobs)
但是我得到了相同的结果。
我认为问题在于循环赋值,而不是plot_grid(),但我看不出我哪里做错了。