我想要找到一个向量
标准的基础R方法并不能完全满足我的需求。例如,使用
另一方面,
我想要的是一个返回“所有x和所有y”的索引的函数。也就是说,它应该返回以下期望的结果:
我们当然可以用R中的循环来实现这个。
但是在大数据上这个速度太慢了(我需要在大数据上多次执行这个操作)
这个速度非常慢,如果我们与
希望在R中有一种快速的方法来做这个?否则,也许Rcpp是一个可行的选择。
x 在另一个查找向量 table 中所有匹配项的索引。table = rep(1:5, each=3)
x = c(2, 5, 2, 6)
标准的基础R方法并不能完全满足我的需求。例如,使用
which(table %in% x),我们只能得到匹配的索引一次,即使x中出现了两次2。which(table %in% x)
# [1] 4 5 6 13 14 15
另一方面,
match 返回每个与 x 匹配的值,但只返回查找表中的第一个索引。match(x, table)
# [1] 4 13 4 NA
我想要的是一个返回“所有x和所有y”的索引的函数。也就是说,它应该返回以下期望的结果:
mymatch(x, table)
# c(4, 5, 6, 13, 14, 15, 4, 5, 6)
我们当然可以用R中的循环来实现这个。
mymatch = function(x, table) {
matches = sapply(x, \(xx) which(table %in% xx))
unlist(matches)
}
mymatch(x, table)
# [1] 4 5 6 13 14 15 4 5 6
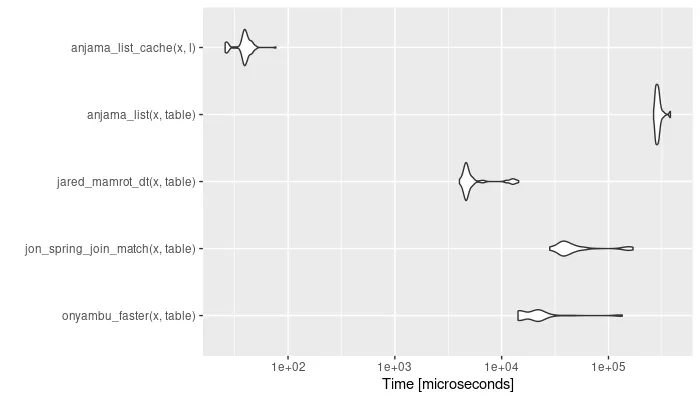
但是在大数据上这个速度太慢了(我需要在大数据上多次执行这个操作)
table = rep(1:1e5, each=10)
x = sample(1:100, 1000, replace = TRUE)
system.time(mymatch(x, table))
# user system elapsed
# 3.279 2.881 6.157
这个速度非常慢,如果我们与
which %in%进行比较的话。system.time(which(table %in% x))
# user system elapsed
# 0.003 0.004 0.008
希望在R中有一种快速的方法来做这个?否则,也许Rcpp是一个可行的选择。

mymatch时,我得到一个10x1000的矩阵,这是期望的输出结构吗? - undefined