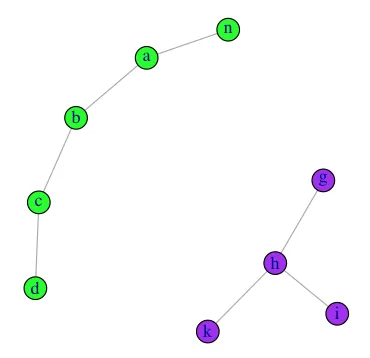
我有一个向量列表如下。
data <- list(v1=c("a", "b", "c"), v2=c("g", "h", "k"),
v3=c("c", "d"), v4=c("n", "a"), v5=c("h", "i"))
我想要实现以下目标:
- 检查任何两个向量之间是否相交
- 如果找到相交的向量,则获取它们的并集
所以期望的输出是:
out <- list(v1=c("a", "b", "c", "d", "n"), v2=c("g", "h", "k", "i"))
我可以通过以下方式获取一组相交集合的并集。
Reduce(union, list(data[[1]], data[[3]], data[[4]]))
Reduce(union, list(data[[2]], data[[5]])
如何首先确定相交向量?有没有一种方法将列表分成相交向量的组列表?
#更新
这里尝试使用data.table。获得所需的结果。但是对于像示例数据集中那样的大列表仍然很慢。
datasets.
data <- sapply(data, function(x) paste(x, collapse=", "))
data <- as.data.frame(data, stringsAsFactors = F)
repeat {
M <- nrow(data)
data <- data.table( data , key = "data" )
data <- data[ , list(dataelement = unique(unlist(strsplit(data , ", " )))), by = list(data)]
data <- data.table(data , key = "dataelement" )
data <- data[, list(data = paste0(sort(unique(unlist(strsplit(data, split=", ")))), collapse=", ")), by = "dataelement"]
data$dataelement <- NULL
data <- unique(data)
N <- nrow(data)
if (M == N)
break
}
data <- strsplit(as.character(data$data) , "," )