我想使用holoviews/hvplot,在一个包含多个唯一数据点的维度上,将几个数据绘制成网格。
考虑这个例子:
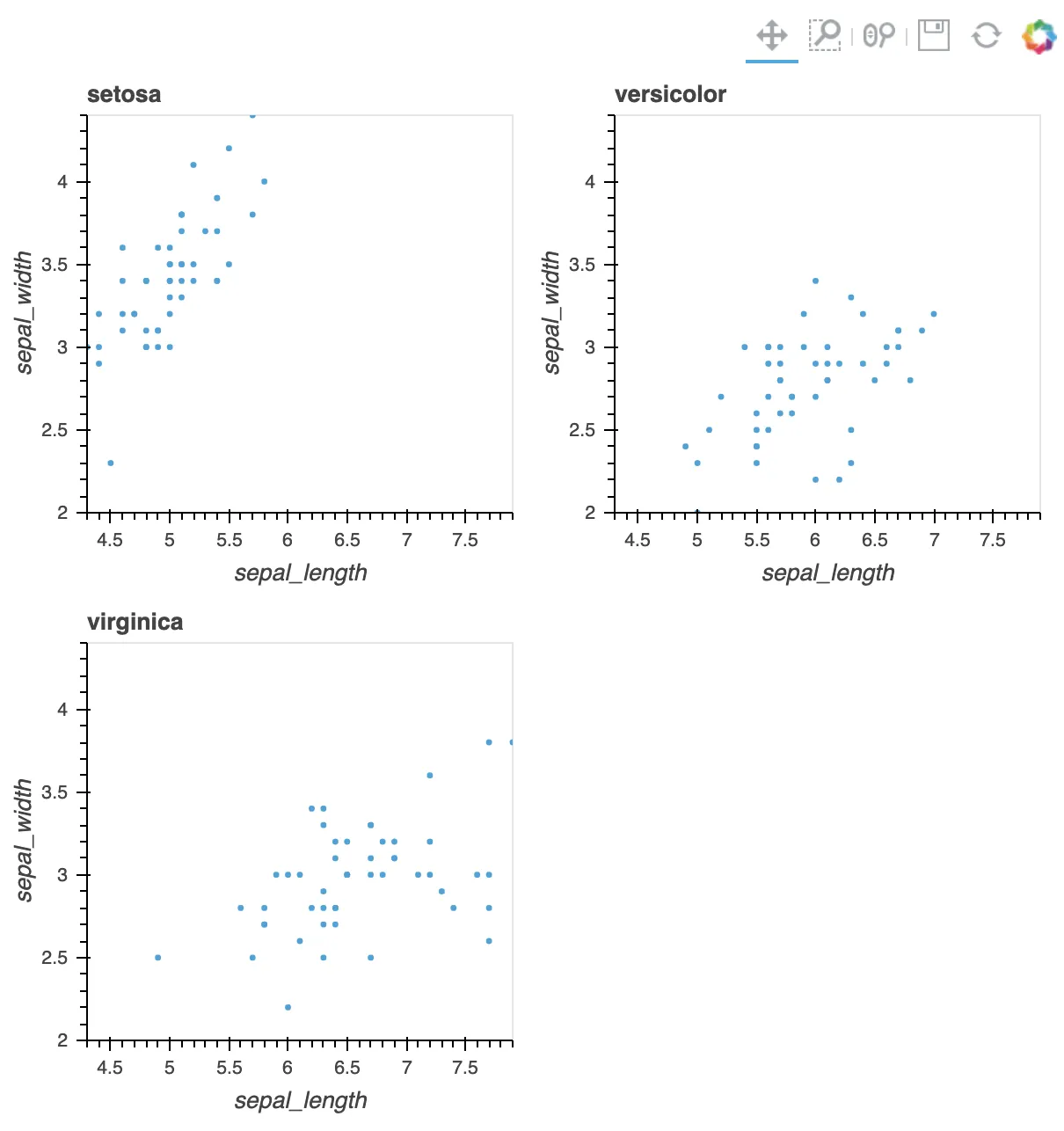
上面的代码根据鸢尾花数据集中物种部分创建了几个图表,结果如下图所示:
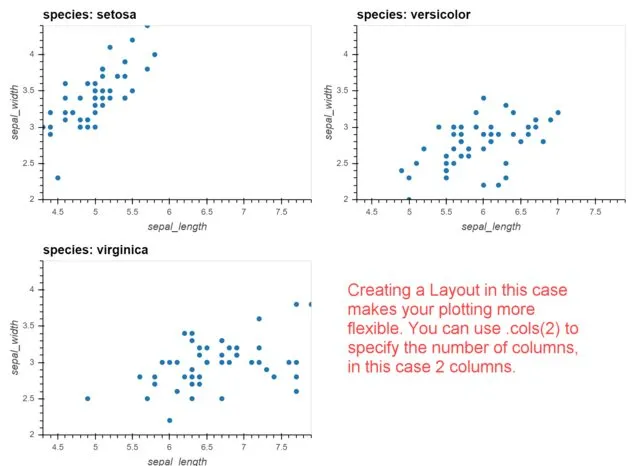
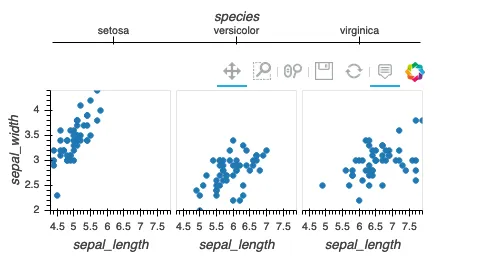 但现在想象一下,如果不是3个不同的物种,而是20个。图表会变得太宽,因此我想在几个图表后换行。但我找不到任何“最大列数”参数。普通网格需要另一列来定义行,但我没有。
请给出任何建议。
但现在想象一下,如果不是3个不同的物种,而是20个。图表会变得太宽,因此我想在几个图表后换行。但我找不到任何“最大列数”参数。普通网格需要另一列来定义行,但我没有。
请给出任何建议。
考虑这个例子:
import seaborn as sns
import hvplot.pandas
iris = sns.load_dataset('iris')
plot = iris.hvplot.scatter(x="sepal_length", y="sepal_width", col="species")
hvplot.show(plot)
上面的代码根据鸢尾花数据集中物种部分创建了几个图表,结果如下图所示:
 但现在想象一下,如果不是3个不同的物种,而是20个。图表会变得太宽,因此我想在几个图表后换行。但我找不到任何“最大列数”参数。普通网格需要另一列来定义行,但我没有。
请给出任何建议。
但现在想象一下,如果不是3个不同的物种,而是20个。图表会变得太宽,因此我想在几个图表后换行。但我找不到任何“最大列数”参数。普通网格需要另一列来定义行,但我没有。
请给出任何建议。