亲爱的众人
问题
我尝试使用nfc、pgirmess、SpatialPack和spdep包计算空间自相关图。然而,我在定义距离的起点和终点时遇到了困难。我只对小距离下的空间自相关性感兴趣,但是这里要使用更小的区间。此外,由于栅格数据集相当大(1.8百万像素),使用这些包但SpatialPack会遇到内存问题。
因此,我尝试自己编写代码,使用raster包中的Moran函数。但是我的结果与其他包的结果有些不同,这可能是因为我的代码中有错误。如果我的代码没有错误,它至少可以帮助其他遇到类似问题的人。
问题
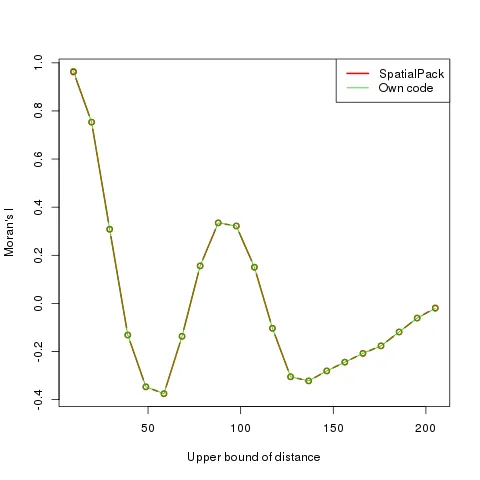
我不确定我的聚焦矩阵是否有误。请问中心像素是否需要被纳入?使用测试数据,我无法展示这些方法之间的差异,但在我的完整数据集上,存在差异,如下面的图片所示。但是,这些区间不完全相同(50m vs. 69m),因此这可能解释了部分差异。但是,在第一个区间,这种解释对我来说似乎不太合理。或者,我的栅格数据集的不规则形状和处理NA的不同方式会引起差异吗?
可运行示例
测试数据
计算测试数据的代码取自http://www.petrkeil.com/?p=1050#comment-416317
# packages used for the data generation
library(raster)
library(vegan) # will be used for PCNM
# empty matrix and spatial coordinates of its cells
side=30
my.mat <- matrix(NA, nrow=side, ncol=side)
x.coord <- rep(1:side, each=side)*5
y.coord <- rep(1:side, times=side)*5
xy <- data.frame(x.coord, y.coord)
# all paiwise euclidean distances between the cells
xy.dist <- dist(xy)
# PCNM axes of the dist. matrix (from 'vegan' package)
pcnm.axes <- pcnm(xy.dist)$vectors
# using 8th PCNM axis as my atificial z variable
z.value <- pcnm.axes[,8]*200 + rnorm(side*side, 0, 1)
# plotting the artificial spatial data
r <- rasterFromXYZ(xyz = cbind(xy,z.value))
plot(r, axes=F)
自己的代码
library(raster)
sp.Corr <- matrix(nrow = 0,ncol = 2)
formerBreak <- 0 #for the first run important
for (i in c(seq(10,200,10))) #Calculate the Morans I for these bins
{
cat(paste0("..",i)) #print the bin, which is currently calculated
w = focalWeight(r,d = i,type = 'circle')
wTemp <- w #temporarily saves the weigtht matrix
if (formerBreak>0) #if it is the second run
{
midpoint <- ceiling(ncol(w)/2) # get the midpoint
w[(midpoint-formerBreak):(midpoint+formerBreak),(midpoint-formerBreak):(midpoint+formerBreak)] <- w[(midpoint-formerBreak):(midpoint+formerBreak),(midpoint-formerBreak):(midpoint+formerBreak)]*(wOld==0)#set the previous focal weights to 0
w <- w*(1/sum(w)) #normalizes the vector to sum the weights to 1
}
wOld <- wTemp #save this weight matrix for the next run
mor <- Moran(r,w = w)
sp.Corr <- rbind(sp.Corr,c(Moran =mor,Distance = i))
formerBreak <- i/res(r)[1]#divides the breaks by the resolution of the raster to be able to translate them to the focal window
}
plot(x=sp.Corr[,2],y = sp.Corr[,1],type = "l",ylab = "Moran's I",xlab="Upper bound of distance")
计算空间相关图的其他方法
library(SpatialPack)
sp.Corr <- summary(modified.ttest(z.value,z.value,coords = xy,nclass = 21))
plot(x=sp.Corr$coef[,1],y = data$coef[,4],type = "l",ylab = "Moran's I",xlab="Upper bound of distance")
library(ncf)
ncf.cor <- correlog(x.coord, y.coord, z.value,increment=10, resamp=1)
plot(ncf.cor)
raster::focalWeight考虑到了这一点,但我认为SpatialPack::modified.ttest没有考虑到(它似乎假设平面坐标系)。 - Robert Hijmans