我有一个患者就诊数据框,想要提取每位患者最早的就诊记录(可以使用顺序就诊ID实现)。我编写的代码可以工作,但我相信使用dplyr可以更有效地执行此任务。您推荐使用什么方法?
以下是4名患者10次就诊的示例:
encounter_ID <- c(1021, 1022, 1013, 1041, 1007, 1002, 1003, 1043, 1085, 1077)
patient_ID <- c(855,721,821,855,423,423,855,721,423,855)
gender <- c(0,0,1,0,1,1,0,0,1,0)
df <- data.frame(encounter_ID, patient_ID, gender)
结果(期望和实际):
encounter_ID patient_ID gender
1003 855 0
1022 721 0
1013 821 1
1002 423 1
我的方法
1)提取唯一病人列表
list.patients <- unique(df$patient_ID)
2) 创建一个空数据框,以接收每个病人第一次就诊的输出结果
one.encounter <- data.frame()
3) 遍历列表中的每个患者,提取他们的第一次就诊记录并填充我们的数据框。
for (i in 1:length(list.patients)) {
one.patient <- df %>% filter(patient_ID==list.patients[i])
one.patient.ordered <- one.patient[order(one.patient$encounter_ID),]
first.encounter <- head(one.patient.ordered, n=1)
one.encounter <- rbind(one.encounter, first.encounter)
}
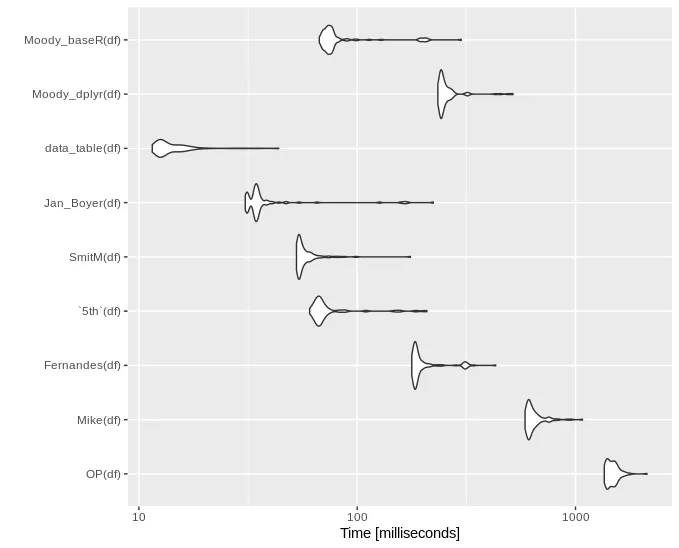
data.table会更快 :D 但是为什么我们的基准测试时间差别这么大呢?你可能在使用 Linux 吗?我的测试是在 Windows 机器上完成的。 - 5thdf的大小问题。你尝试过使用我生成的相同数据进行基准测试吗?我在使用 Linux。 - markus