概括: 处理多个等距点和维度诅咒将是比仅仅找到这些点更大的问题。剧透:有一个惊喜结局。
我认为这是一个有趣的问题,但我对某些答案感到困惑。我认为这部分原因是由于提供的草图。您无疑已经注意到答案看起来相似--2D,带有聚类--即使您指出需要更广泛的范围。因为其他人最终也会看到这个问题,所以我会慢慢地解释我的思路,请在前面耐心等待。
从一个简化的例子开始是有意义的,以查看是否可以用易于理解的数据和线性2D模型来推广解决方案。
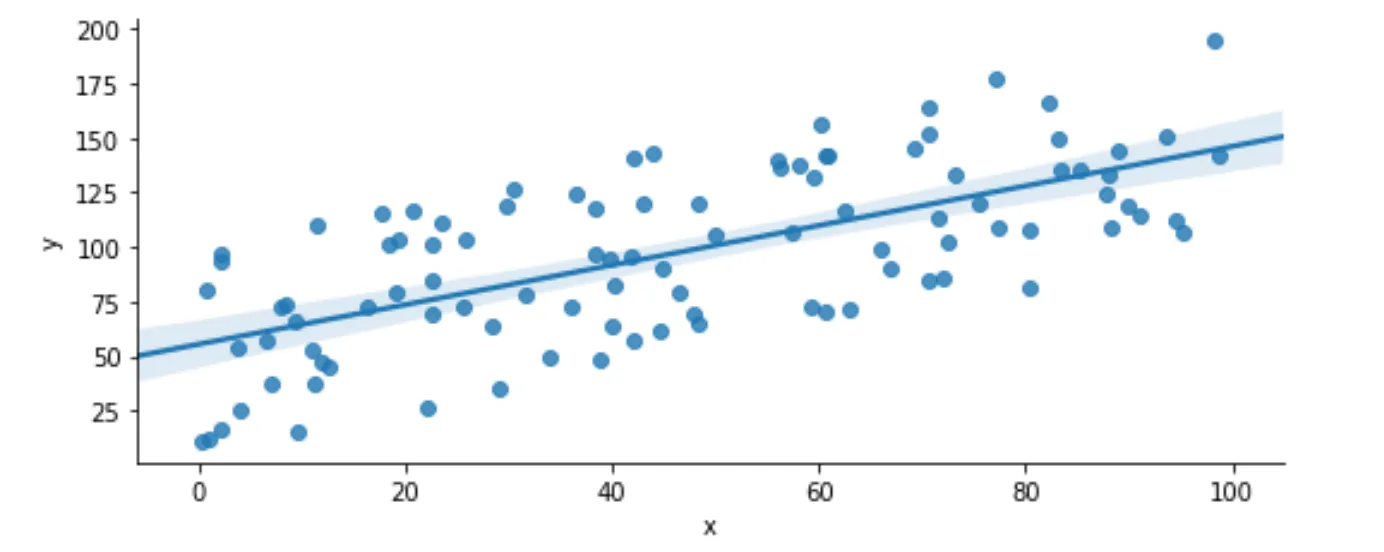 但我们不需要计算所有的距离。我们只需要极端值。因此,我们可以选择一些最高和最低值:
但我们不需要计算所有的距离。我们只需要极端值。因此,我们可以选择一些最高和最低值:
right = lin_2_D.nlargest(8, ['x'])
left = lin_2_D.nsmallest(8, ['x'])
graph = sns.scatterplot(x="x", y="y", data=lin_2_D, color = 'gray', marker = '+', alpha = .4)
sns.scatterplot(x = right['x'], y = right['y'], color = 'red')
sns.scatterplot(x = left['x'], y = left['y'], color = 'green')
fig = graph.figure
fig.set_size_inches(8,3)
我们目前所拥有的是:在100个点中,我们已经消除了需要计算84个点之间距离的需求。对于剩下的点,我们可以通过将结果排序并检查其与其他点之间的距离来进一步减少这一需求。
你可以想象一个情况,即您有几个数据点远离趋势线,可以通过取最大或最小的y值来捕捉,所有这些看起来都像Walter Tross的顶部图表。再添加几个额外的聚类,就会得到他底部的图表,看起来我们正在阐述同样的观点。
停留在这里的问题在于,您提到的要求是需要解决任意维度的问题。
不幸的是,我们遇到了四个挑战:
挑战1:随着维度的增加,您可能会遇到许多情况,在寻找中点时存在多个解决方案。因此,您正在寻找k个最远的点,但存在大量同样有效的可能解决方案,并且没有优先考虑它们的方法。以下是两个超级简单的例子说明这一点:
在这里,我们只有四个点和两个维度。你真的找不到比这更简单的了,对吗?从红色到绿色的距离微不足道。但是试着找到下一个最远的点,你会发现两个黑点都与红点和绿点等距离。想象一下,如果你使用第一个图表来找到最远的六个点,你可能会有20个或更多个点都等距离。
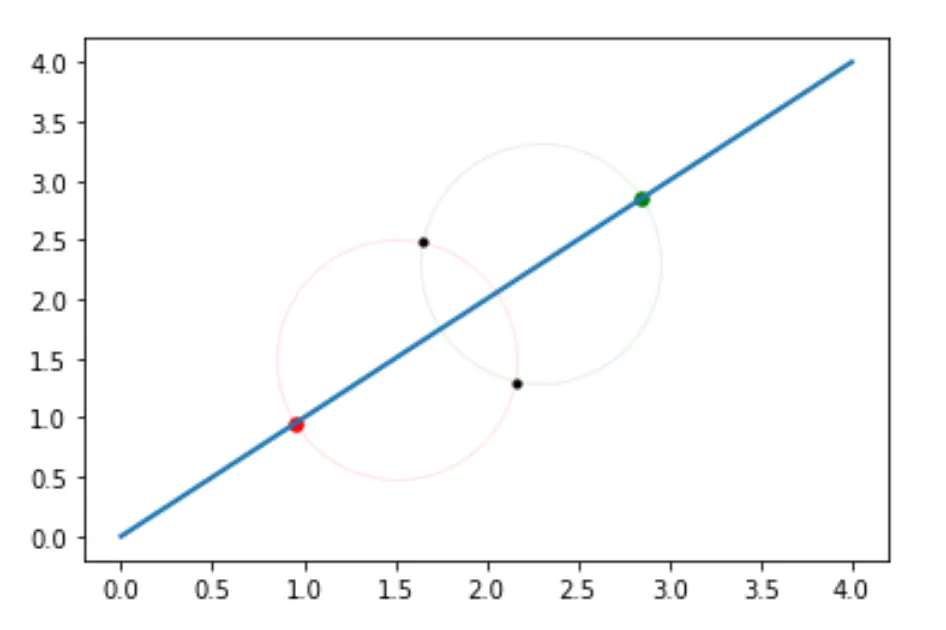
编辑:我刚注意到红色和绿色的点是在它们的圆的边缘而不是中心,我稍后会更新,但重点是相同的。
B)这很容易想象:想象一下D&D四面体骰子。在三维空间中有四个数据点,所有点距离相等,因此被称为三角形金字塔。如果你正在寻找最接近的两个点,那么哪两个?你有4个选择2(也就是6)种可能的组合。摆脱有效解决方案可能会有些问题,因为你不可避免地会面临问题,比如“为什么我们要摆脱这些而不是这一个?”
挑战2:
维度诅咒。说的够清楚了。
挑战3 维度诅咒的复仇 因为你正在寻找最远的点,所以你必须对每个点进行x、y、z...n坐标或者你必须插值它们。现在,你的数据集更大,速度更慢。
挑战4 因为你正在寻找最远的点,因此降维技术如岭回归和套索将没有用处。
那么,对此该怎么办呢?
什么也不做。
等待。什么?!?
并非真正地、确切地和字面上的“什么也不做”。而是采用一种简单的启发式方法,这种方法易于理解和计算。保罗·凯宁表达得很好:
直观地说,当情况足够复杂或不确定时,只有最简单的方法才有效。然而,基于这些稳健适用技术的常识启发式方法可以产生几乎肯定是最优的结果。
在这种情况下,你没有遭受维数灾难,而是拥有维数祝福。的确,你有很多点,当你寻找其他等距点(k)时,它们会按线性比例增加,但空间的总维度体积将增加到维度的幂次方。你所关心的k个最远点与总点数相比微不足道。甚至k^2随着维数的增加也变得微不足道。
现在,如果你的维数较低,我会选择它们作为解决方案(除了使用嵌套for循环的解决方案……在NumPy或Pandas中)。
如果我处在你的位置,我会考虑我已经在其他答案中编写了可以用作基础的代码,并可能会想知道为什么我应该相信这个比其他答案更好,除了它提供了一个思考这个主题的框架。当然,应该有一些数学知识,也许是一些重要人物说同样的话。
让我引用
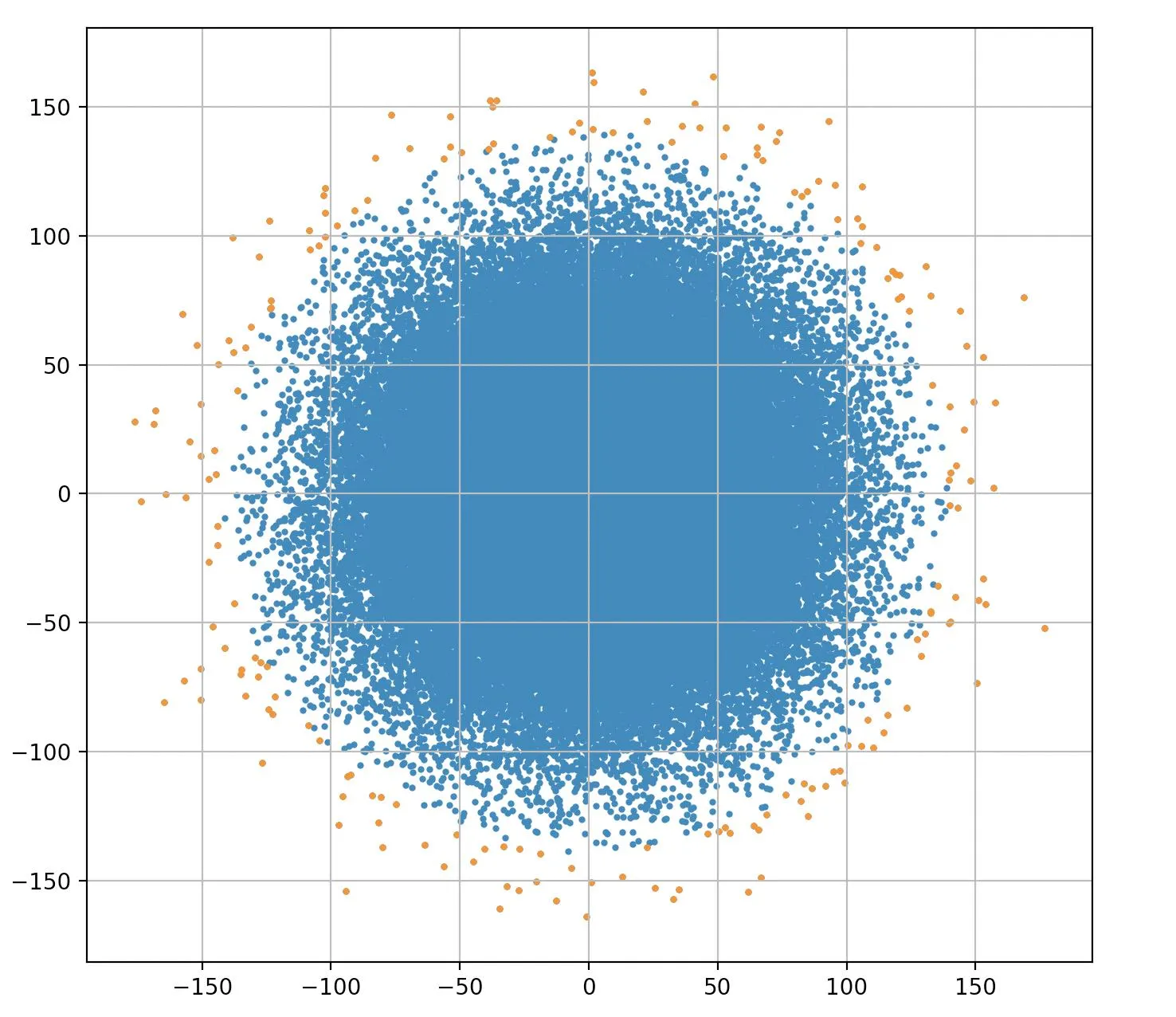
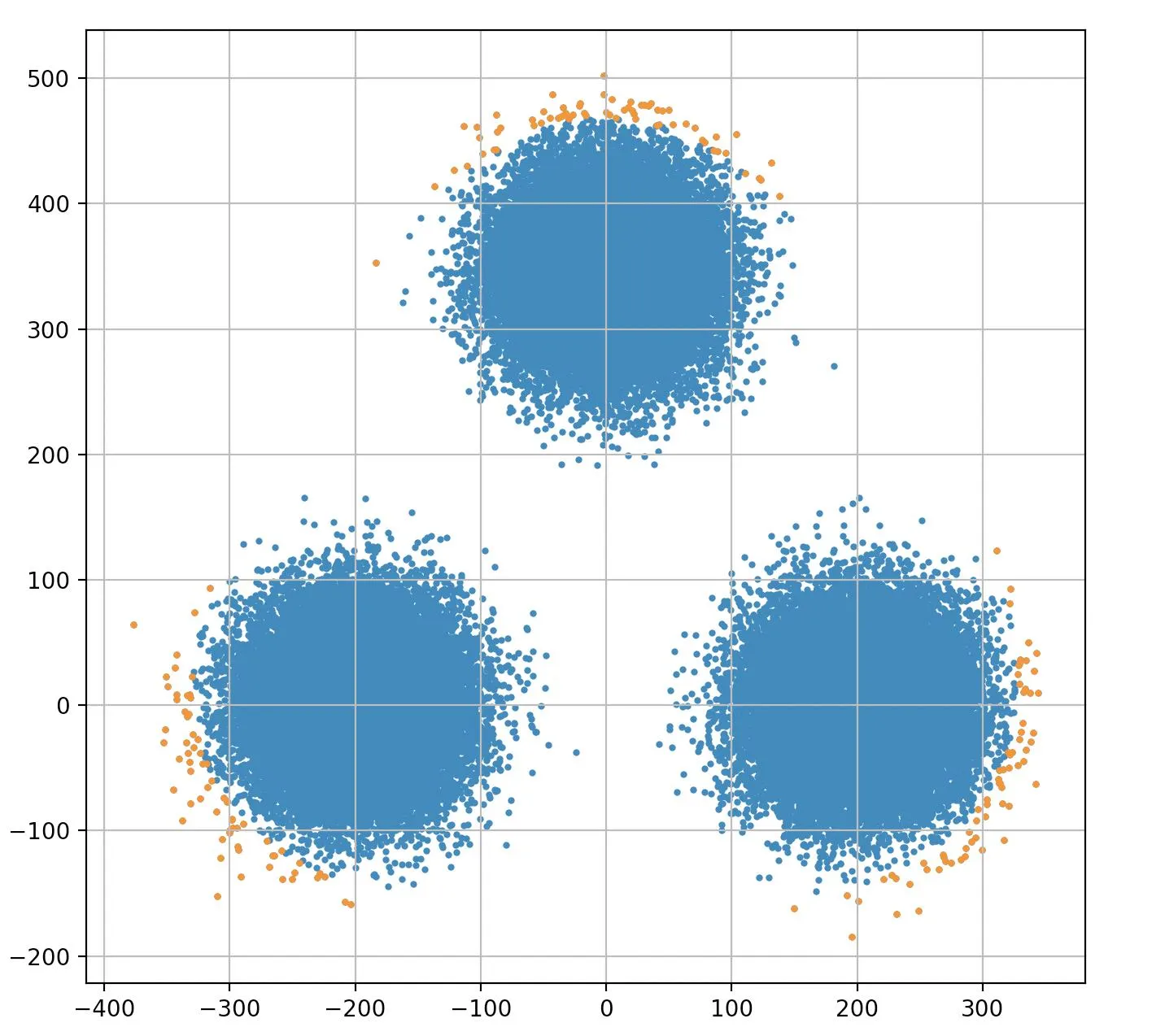
《计算机控制和信号处理的计算密集方法》第18章和通过类比进行扩展的论证,其中包含一些繁重的数学知识。从上面的图表(边缘带有彩色点的图表)可以看出,中心被移除了,特别是如果你遵循了去除极端y值的想法。就像你把一个气球放在一个盒子里一样。你也可以把球放在一个立方体里。将其升级到多个维度,你就有了一个超球体在一个超立方体中。你可以在这里
阅读更多关于这种关系的信息。
最后,让我们来谈谈一种启发式方法:
选择每个维度中具有最大或最小值的点。当/如果你用完了这些点,就选择靠近这些值的点,如果在最小/最大值处没有一个点。本质上,您正在选择一个框的角落。对于二维图形,您有四个点,对于三维图形,您有一个框的8个角(2^3)。
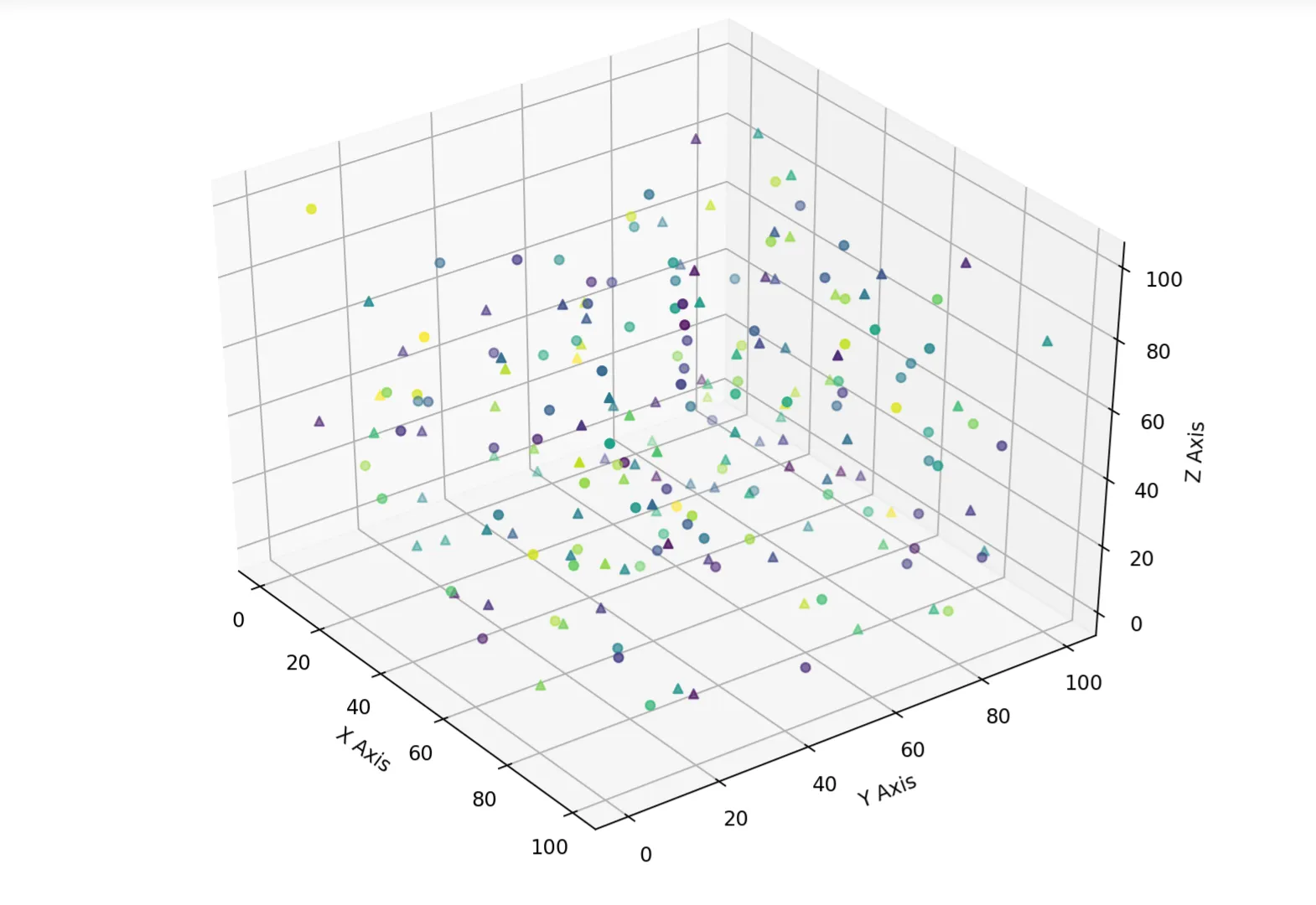
更准确地说,这将是一个4D或5D(取决于您如何分配标记形状和颜色)投影到3D。但您可以轻松地看到此数据云给出了完整的尺寸范围。
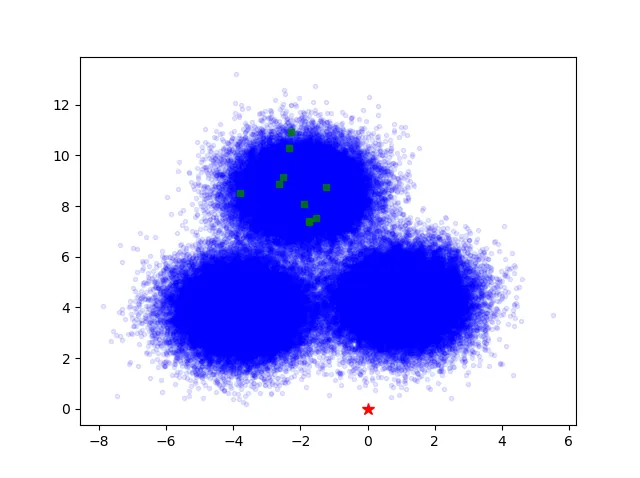
这里是一个快速的学习检查; 为了简单起见,忽略颜色/形状方面:你可以直观地判断,在决定可能稍微靠近的点之前,你最多可以处理 k 个点而没有问题。如果你有一个k < 2D,你可以看到你可能需要随机选择。如果你添加了另一个点,你可以看到它(k +1)会在一个质心中。所以这是检查:如果你有更多的点,它们会在哪里?我想我必须把这放在底部 - markdown的限制。
因此,对于一个6D数据云,小于64(实际上是65,我们马上就会看到)点的值相当容易。但是......
如果您没有数据云,而是有具有线性关系的数据,则将获得2^(D-1)个点。因此,在线性2D空间中,您有一条直线,在线性3D空间中,您将有一个平面。然后是菱形等等,即使您的形状是弯曲的,这也是正确的。我不想自己制作这个图表,而是使用来自Inversion Labs的优秀帖子
Best-fit Surfaces for 3D Data中的图表。
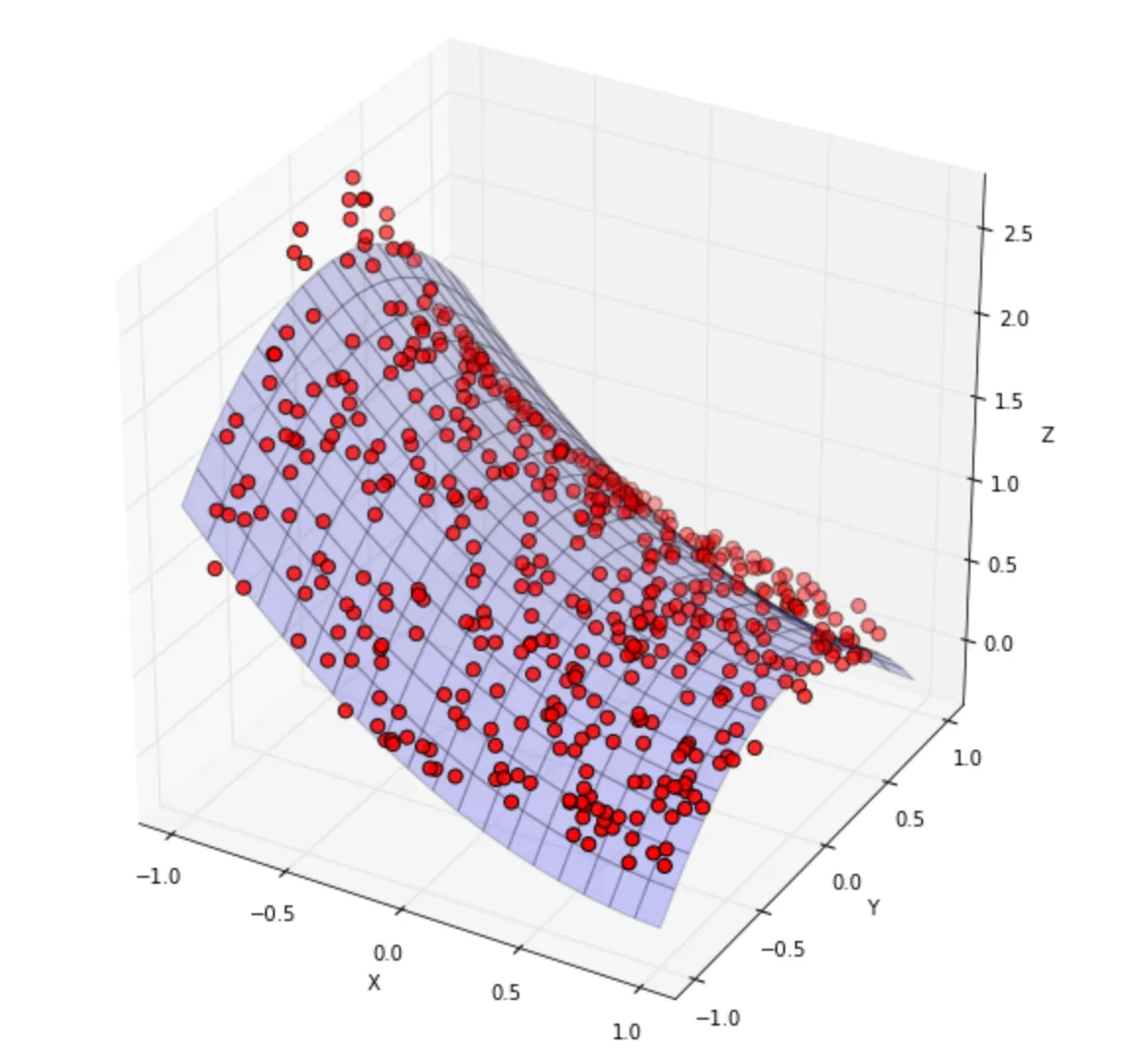
如果点数k小于2^D,你需要一个过程来决定哪些点不使用。 线性判别分析 应该在你的备选名单上。尽管如此,你可以通过随机选择一个点来满足解决方案。
对于一个额外的点(k = 1 + 2^D),你要寻找一个距离边界空间中心最近的点。
当k > 2^D时,可能的解决方案将呈阶乘而非几何级数增长。这可能看起来不直观,所以让我们回到两个圆的例子。在二维空间中,只有两个点可能成为等距离候选点。但如果是三维空间并围绕着一条线旋转这些点,则现在任何在环状区域内的点都可以作为k的解决方案。对于三维例子,它们将是球体。从那里开始就是超球体(n-球体)。再次,2^D的比例。
最后一件事:如果您还不熟悉
xarray,那么您应该认真考虑一下。
希望所有这些都有所帮助,也希望您会阅读这些链接。它们值得花时间去看。
*它将具有相同的形状,位于中心位置,顶点位于1/3处。就像有27个六面骰子形状的巨大立方体一样。每个顶点(或最近的点)将确定解决方案。您的原始k+1也必须重新定位。因此,您将选择8个顶点中的2个。最后一个问题:是否值得计算这些点之间的距离(请记住对角线略长于边缘),然后将它们与原始的2^D点进行比较?坦率地说,不值得。牺牲这个解决方案。
 or this
or this