这是用Python而不是R编写的,但jbaums说可以。
以下是我的贡献,请参见源代码中的注释以了解关键部分的说明。
我仍在努力分析解决方案,确定深度为t和S个样本的树可能组合的数量c,以便改进combs函数。也许有人有这个解决方案吗?
这真的是现在的瓶颈。
从深度为16的树中抽取100个节点,在我的笔记本电脑上大约需要8毫秒。不是第一次,但随着抽样次数的增加,它会变得更快,因为combBuffer被填满了。
import random
class Tree(object):
"""
:param level: The distance of this node from the root.
:type level: int
:param parent: This trees parent node
:type parent: Tree
:param isleft: Determines if this is a left or a right child node. Can be
omitted if this is the root node.
:type isleft: bool
A binary tree representing possible strings which match r'[01]{1,n}'. Its
purpose is to be able to sample n of its nodes where none of the sampled
nodes' ids is a prefix for another one.
It is possible to change Tree.maxdepth and then reuse the root. All
children are created ON DEMAND, which means everything is lazily evaluated.
If the Tree gets too big anyway, you can call 'prune' on any node to delete
its children.
>>> t = Tree()
>>> t.sample(8, toString=True, depth=3)
['111', '110', '101', '100', '011', '010', '001', '000']
>>> Tree.maxdepth = 2
>>> t.sample(4, toString=True)
['11', '10', '01', '00']
"""
maxdepth = 10
_combBuffer = {}
def __init__(self, level=0, parent=None, isleft=None):
self.parent = parent
self.level = level
self.isleft = isleft
self._left = None
self._right = None
@classmethod
def setMaxdepth(cls, depth):
"""
:param depth: The new depth
:type depth: int
Sets the maxdepth of the Trees. This basically is the depth of the root
node.
"""
if cls.maxdepth == depth:
return
cls.maxdepth = depth
@property
def left(self):
"""This tree's left child, 'None' if this is a leave node"""
if self.depth == 0:
return None
if self._left is None:
self._left = Tree(self.level+1, self, True)
return self._left
@property
def right(self):
"""This tree's right child, 'None' if this is a leave node"""
if self.depth == 0:
return None
if self._right is None:
self._right = Tree(self.level+1, self, False)
return self._right
@property
def depth(self):
"""
This tree's depth. (maxdepth-level)
"""
return self.maxdepth-self.level
@property
def id(self):
"""
This tree's id, string of '0's and '1's equal to the path from the root
to this subtree. Where '1' means going left and '0' means going right.
"""
if self.level == 0:
return ''
return self.parent.id + ('1' if self.isleft else '0')
@property
def leaves(self):
"""
The amount of leave nodes, this tree has. (2**depth)
"""
return 2**self.depth
def __str__(self):
return self.id
def __len__(self):
return 2*self.leaves-1
def prune(self):
"""
Recursively prune this tree's children.
"""
if self._left is not None:
self._left.prune()
self._left.parent = None
self._left = None
if self._right is not None:
self._right.prune()
self._right.parent = None
self._right = None
def combs(self, n):
"""
:param n: The amount of samples to be taken from this tree
:type n: int
:returns: The amount of possible combinations to choose n samples from
this tree
Determines recursively the amount of combinations of n nodes to be
sampled from this tree.
Subsequent calls with same n on trees with same depth will return the
result from the previous computation rather than computing it again.
>>> t = Tree()
>>> Tree.maxdepth = 4
>>> t.combs(16)
1
>>> Tree.maxdepth = 3
>>> t.combs(6)
58
"""
key = (self.depth, n)
if key not in Tree._combBuffer:
leaves = self.leaves
if n < 0:
N = 0
elif n == 0 or self.depth == 0 or n == leaves:
N = 1
elif n == 1:
return (2*leaves-1)
else:
if n > leaves/2:
nMin = n-leaves/2
else:
nMin = 0
free = n-2*nMin
N = 0
for addLeft in range(0, free+1):
nLeft = nMin + addLeft
nRight = n - nLeft
N += self.left.combs(nLeft)*self.right.combs(nRight)
Tree._combBuffer[key] = N
return N
return Tree._combBuffer[key]
def sample(self, n, toString=False, depth=None):
"""
:param n: How may samples to take from this tree
:type n: int
:param toString: If 'True' result will direclty be turned into a list
of strings
:type toString: bool
:param depth: If not None, will overwrite Tree.maxdepth
:type depth: int or None
:returns: List of n nodes sampled from this tree
:throws ValueError: when n is invalid
Takes n random samples from this tree where none of the sample's ids is
a prefix for another one's.
For an example see Tree's docstring.
"""
if depth is not None:
Tree.setMaxdepth(depth)
if toString:
return [str(e) for e in self.sample(n)]
if n < 0:
raise ValueError('Negative sample size is not possible!')
if n == 0:
return []
leaves = self.leaves
if n > leaves:
raise ValueError(('Cannot sample {} nodes, with only {} ' +
'leaves!').format(n, leaves))
if n == 1 and self.level > 0:
P_root = random.randint(0, len(self)-1)
if P_root == 0:
return [self]
else:
if random.random() < 0.5:
return self.right.sample(1)
else:
return self.left.sample(1)
if n > leaves/2:
nMin = n-leaves/2
else:
nMin = 0
free = n-2*nMin
r = random.randint(0, self.combs(n)-1)
p = 0
for addLeft in xrange(0, free+1):
nLeft = nMin + addLeft
nRight = n - nLeft
p += (self.left.combs(nLeft) * self.right.combs(nRight))
if r < p:
return self.left.sample(nLeft) + self.right.sample(nRight)
assert False, ('Something really strange happend, p did not sum up ' +
'to combs or r was too big')
def main():
"""
Do a microbenchmark.
"""
import timeit
i = 1
main.t = Tree()
template = ' {:>2} {:>5} {:>4} {:<5}'
print(template.format('i', 'depth', 'n', 'time (ms)'))
N = 100
for depth in [4, 8, 15, 16, 17, 18]:
for n in [10, 50, 100, 150]:
if n > 2**depth:
time = '--'
else:
time = timeit.timeit(
'main.t.sample({}, depth={})'.format(n, depth), setup=
'from __main__ import main', number=N)*1000./N
print(template.format(i, depth, n, time))
i += 1
if __name__ == "__main__":
main()
基准测试输出:
i depth n time (ms)
1 4 10 0.182511806488
2 4 50 --
3 4 100 --
4 4 150 --
5 8 10 0.397620201111
6 8 50 1.66054964066
7 8 100 2.90236949921
8 8 150 3.48146915436
9 15 10 0.804011821747
10 15 50 3.7428188324
11 15 100 7.34910964966
12 15 150 10.8230614662
13 16 10 0.804491043091
14 16 50 3.66818904877
15 16 100 7.09567070007
16 16 150 10.404779911
17 17 10 0.865840911865
18 17 50 3.9999294281
19 17 100 7.70257949829
20 17 150 11.3758206367
21 18 10 0.915451049805
22 18 50 4.22935962677
23 18 100 8.22361946106
24 18 150 12.2081303596
从深度为10的树中抽取10个大小为10的样本:
['1111010111', '1110111010', '1010111010', '011110010', '0111100001', '011101110', '01110010', '01001111', '0001000100', '000001010']
['110', '0110101110', '0110001100', '0011110', '0001111011', '0001100010', '0001100001', '0001100000', '0000011010', '0000001111']
['11010000', '1011111101', '1010001101', '1001110001', '1001100110', '10001110', '011111110', '011001100', '0101110000', '001110101']
['11111101', '110111', '110110111', '1101010101', '1101001011', '1001001100', '100100010', '0100001010', '0100000111', '0010010110']
['111101000', '1110111101', '1101101', '1101000000', '1011110001', '0111111101', '01101011', '011010011', '01100010', '0101100110']
['1111110001', '11000110', '1100010100', '101010000', '1010010001', '100011001', '100000110', '0100001111', '001101100', '0001101101']
['111110010', '1110100', '1101000011', '101101', '101000101', '1000001010', '0111100', '0101010011', '0101000110', '000100111']
['111100111', '1110001110', '1100111111', '1100110010', '11000110', '1011111111', '0111111', '0110000100', '0100011', '0010110111']
['1101011010', '1011111', '1011100100', '1010000010', '10010', '1000010100', '0111011111', '01010101', '001101', '000101100']
['111111110', '111101001', '1110111011', '111011011', '1001011101', '1000010100', '0111010101', '010100110', '0100001101', '0010000000']
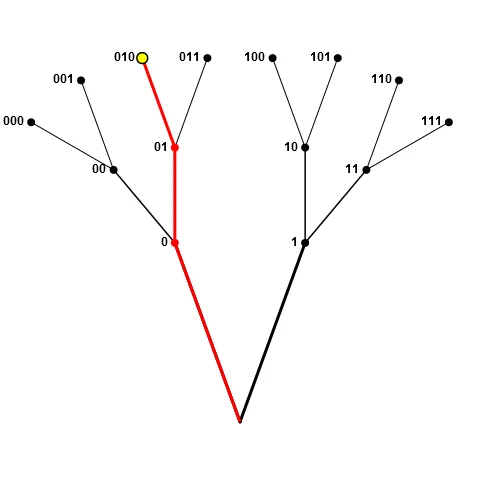
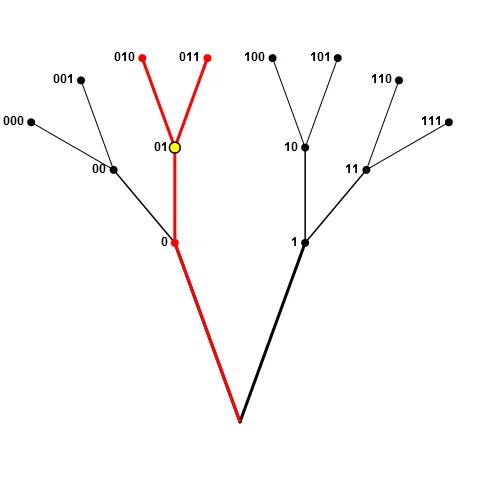
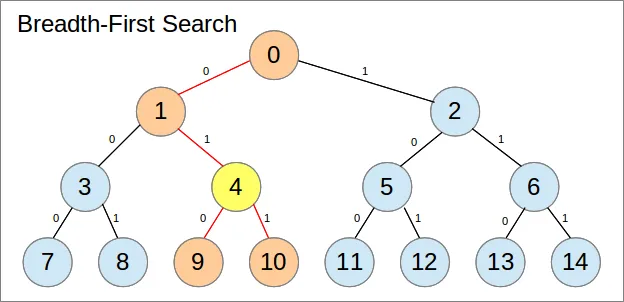 作为一个数组,它占用的内存较少(没有更多指针),所使用的内存是连续的(对于缓存很好),并且可以完全放在堆栈上而不是堆(假设您的语言给您选择)。当然,这里有一些条件,特别是数组的大小。我稍后会回到这个问题。
作为一个数组,它占用的内存较少(没有更多指针),所使用的内存是连续的(对于缓存很好),并且可以完全放在堆栈上而不是堆(假设您的语言给您选择)。当然,这里有一些条件,特别是数组的大小。我稍后会回到这个问题。