我将尝试在rjags中建模一个三级嵌套线性混合效应模型(三级:多个个体在多个组内有多个观察结果)。 在这些组中存在独特的个体集合。
相当的lme4模型为:
运行模型
相当的lme4模型为:
lmer(yN ~ x + (1 |group/indiv), data=qq)
或者
lmer(yN ~ x + (1 |group) + (1|indiv), data=qq)
我的问题是:请问如何在rjags中编写这个模型。
以下是我尝试使用rjags编写的代码,它可以编译和执行,但是个体水平的随机效应似乎被惩罚得太多了——足以表明它编码错误。
st <- "
model {
for(i in 1:n){
mu[i] <- beta[1] + b1[ind[i]] + b2[group[i]] + beta[2]* x[i]
y[i] ~ dnorm(mu[i], tau)
}
for(i in 1:2){ beta[i] ~ dnorm(0, 0.0001) }
tau ~ dgamma(0.01, 0.01)
sigma <- sqrt(1/tau)
# hierarchical model
for (i in 1:nInd) { b1[i] ~ dnorm(0, tau0) }
for (i in 1:nGrp) { b2[i] ~ dnorm(0, tau1) }
tau0 ~ dgamma(0.001, 0.001)
sigma0 <- sqrt(1/tau0)
tau1 ~ dgamma(0.001, 0.001)
sigma1 <- sqrt(1/tau1)
}
"
运行模型
library(rjags)
mod <- jags.model( textConnection(st),
data=list(y=qq$yN,
x=qq$x,
ind=qq$indiv,
group=qq$group,
n=nrow(qq),
nInd=length(unique(qq$indiv)),
nGrp=length(unique(qq$group))),
n.adapt=1e6,
inits=list(.RNG.seed=1,
.RNG.name="base::Wichmann-Hill")
)
mod <- coda.samples(mod,
variable.names=c("beta","b1", "b2", "sigma", "sigma0", "sigma1"),
n.iter=1e6,
thin=5)
summary(mod)
qq <- structure(list(yN = c(3.51, 5.13, 5.2, 7.46, 5.64, 5.14, 6.84,
7.19, 7.77, 6, 10.97, 9.75, 5.43, 1.11, 10.31, 5.3, 4.52, 4.62,
3.97, 4.31, 8.2, 7.24, 6.75, 0, 7.77, 4.25, 5.29, 2.46, 4.3,
6.67, 8.72, 7.52, 6.12, 6.02, 1.48, 4.65, 7.52, 5.88, 6.06, 5.27,
6.04, 5.36, 7.34, 6.39, 2.84, 3.95, 8.07, 7.22, 4.78, 9.92, 5.85,
2.75, 6.34, 2.62, 7.3, 15.45, 5, 1.52, 8.3, 6.25, 16.32, 5.67,
8.55, 5.72, 2.8, 6.06, 1.3, 11.74, 7.02, 12.85, 6.46, 3.68, 8.48,
0.28, 0.92), x = c(-0.63, 0.18, -0.84, 1.6, 0.33, -0.82, 0.49,
0.74, 0.58, -0.31, 1.51, 0.39, -0.62, -2.21, 1.12, -0.04, -0.02,
0.94, 0.82, 0.59, 0.92, 0.78, 0.07, -1.99, 0.62, -0.06, -0.16,
-1.47, -0.48, 0.42, 1.36, -0.1, 0.39, -0.05, -1.38, -0.41, -0.39,
-0.06, 1.1, 0.76, -0.16, -0.25, 0.7, 0.56, -0.69, -0.71, 0.36,
0.77, -0.11, 0.88, 0.4, -0.61, 0.34, -1.13, 1.43, 1.98, -0.37,
-1.04, 0.57, -0.14, 2.4, -0.04, 0.69, 0.03, -0.74, 0.19, -1.8,
1.47, 0.15, 2.17, 0.48, -0.71, 0.61, -0.93, -1.25), indiv = structure(c(1L,
1L, 1L, 1L, 1L, 2L, 2L, 2L, 2L, 2L, 3L, 3L, 3L, 3L, 3L, 4L, 4L,
4L, 4L, 4L, 5L, 5L, 5L, 5L, 5L, 6L, 6L, 6L, 6L, 6L, 7L, 7L, 7L,
7L, 7L, 8L, 8L, 8L, 8L, 8L, 9L, 9L, 9L, 9L, 9L, 10L, 10L, 10L,
10L, 10L, 11L, 11L, 11L, 11L, 11L, 12L, 12L, 12L, 12L, 12L, 13L,
13L, 13L, 13L, 13L, 14L, 14L, 14L, 14L, 14L, 15L, 15L, 15L, 15L,
15L), .Label = c("a", "b", "c", "d", "e", "f", "g", "h", "i",
"j", "k", "l", "m", "n", "o"), class = "factor"), group = structure(c(1L,
1L, 1L, 1L, 1L, 1L, 1L, 1L, 1L, 1L, 1L, 1L, 1L, 1L, 1L, 2L, 2L,
2L, 2L, 2L, 2L, 2L, 2L, 2L, 2L, 2L, 2L, 2L, 2L, 2L, 3L, 3L, 3L,
3L, 3L, 3L, 3L, 3L, 3L, 3L, 3L, 3L, 3L, 3L, 3L, 4L, 4L, 4L, 4L,
4L, 4L, 4L, 4L, 4L, 4L, 4L, 4L, 4L, 4L, 4L, 5L, 5L, 5L, 5L, 5L,
5L, 5L, 5L, 5L, 5L, 5L, 5L, 5L, 5L, 5L), .Label = c("A", "B",
"C", "D", "E"), class = "factor")), .Names = c("yN", "x", "indiv",
"group"), row.names = c(NA, -75L), class = "data.frame")
在一个类似的例子中,可以通过创建交互变量并将其用作分组变量来考虑数据的嵌套结构(与先前的唯一集合组内相似)。
data(Pastes, package="lme4")
lmer(strength ~ 1 + (1|batch/cask), data=Pastes)
lmer(strength ~ 1 + (1|batch) + (1|batch:cask), data=Pastes) # equivalent
如何在 jags 中编写此代码,是否可以在不创建中间交互变量的情况下完成?
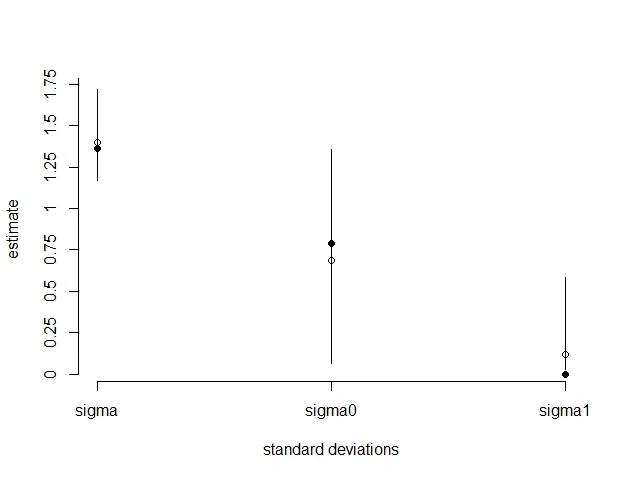
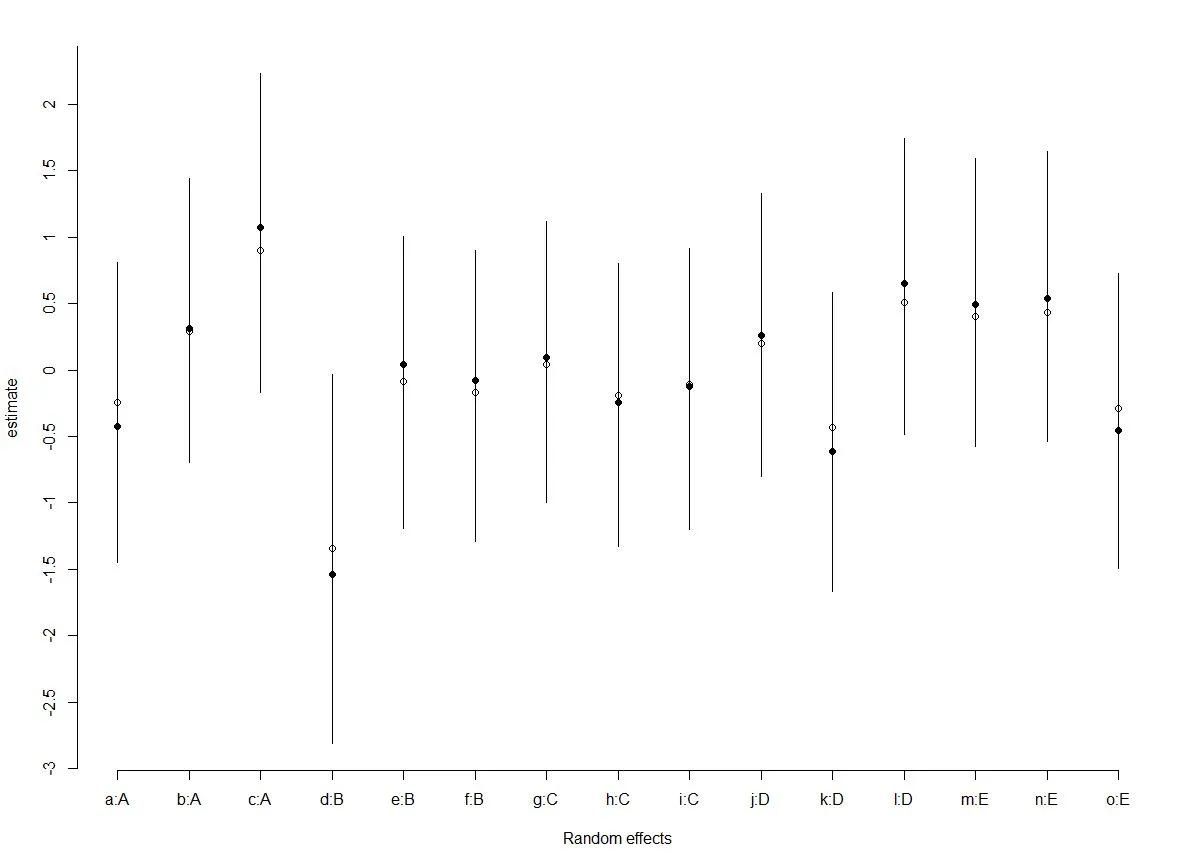
beta [1]添加回到每个b1中,则估计值应该非常接近lme4中报告的值。 - mfidinob预测应该与ranef(lme4model)相同。 - user2957945lmer模型中的任何一个,定义或返回的随机项之间没有相关性(如果我理解正确)。 - user2957945