我一直在尝试测试数据框中最有效的替换NA值的方法。
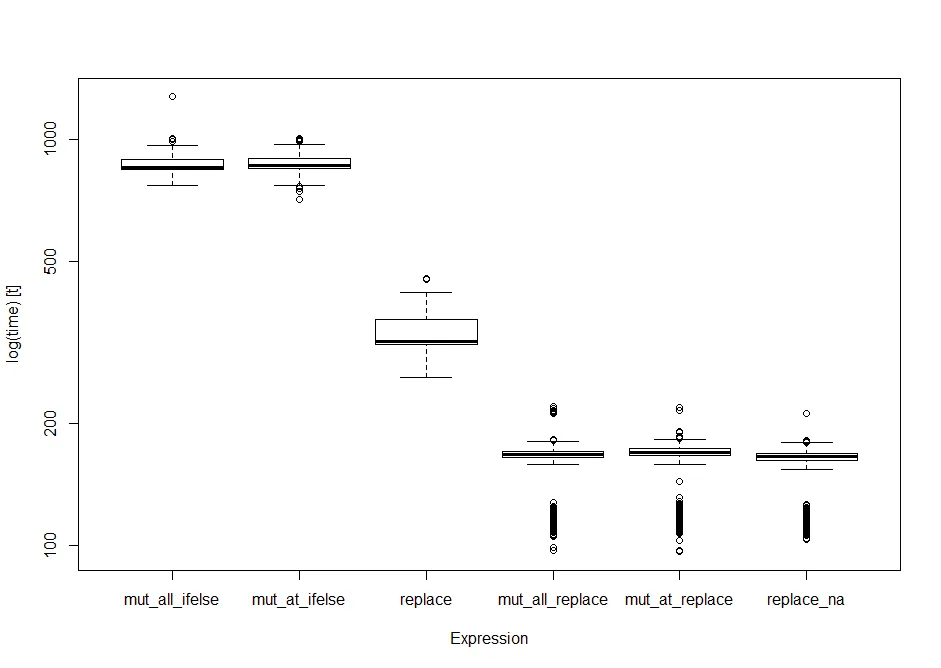
我首先比较了针对1百万行、12列数据集的NA值和0值替换解决方案的效率。将所有管道可用的方案放入microbenchmark函数中,我得到了如下结果。
问题1:有没有办法在benchmark函数内测试子集左赋值语句(例如:df1[is.na(df1)] <- 0)?
library(dplyr)
library(tidyr)
library(microbenchmark)
set.seed(24)
df1 <- as.data.frame(matrix(sample(c(NA, 1:5), 1e6 *12, replace=TRUE),
dimnames = list(NULL, paste0("var", 1:12)), ncol=12))
op <- microbenchmark(
mut_all_ifelse = df1 %>% mutate_all(funs(ifelse(is.na(.), 0, .))),
mut_at_ifelse = df1 %>% mutate_at(funs(ifelse(is.na(.), 0, .)), .cols = c(1:12)),
# df1[is.na(df1)] <- 0 would sit here, but I can't make it work inside this function
replace = df1 %>% replace(., is.na(.), 0),
mut_all_replace = df1 %>% mutate_all(funs(replace(., is.na(.), 0))),
mut_at_replace = df1 %>% mutate_at(funs(replace(., is.na(.), 0)), .cols = c(1:12)),
replace_na = df1 %>% replace_na(list(var1 = 0, var2 = 0, var3 = 0, var4 = 0, var5 = 0, var6 = 0, var7 = 0, var8 = 0, var9 = 0, var10 = 0, var11 = 0, var12 = 0)),
times = 1000L
)
print(op) #standard data frame of the output
Unit: milliseconds
expr min lq mean median uq max neval
mut_all_ifelse 769.87848 844.5565 871.2476 856.0941 895.4545 1274.5610 1000
mut_at_ifelse 713.48399 847.0322 875.9433 861.3224 899.7102 1006.6767 1000
replace 258.85697 311.9708 334.2291 317.3889 360.6112 455.7596 1000
mut_all_replace 96.81479 164.1745 160.6151 167.5426 170.5497 219.5013 1000
mut_at_replace 96.23975 166.0804 161.9302 169.3984 172.7442 219.0359 1000
replace_na 103.04600 161.2746 156.7804 165.1649 168.3683 210.9531 1000
boxplot(op) #boxplot of output
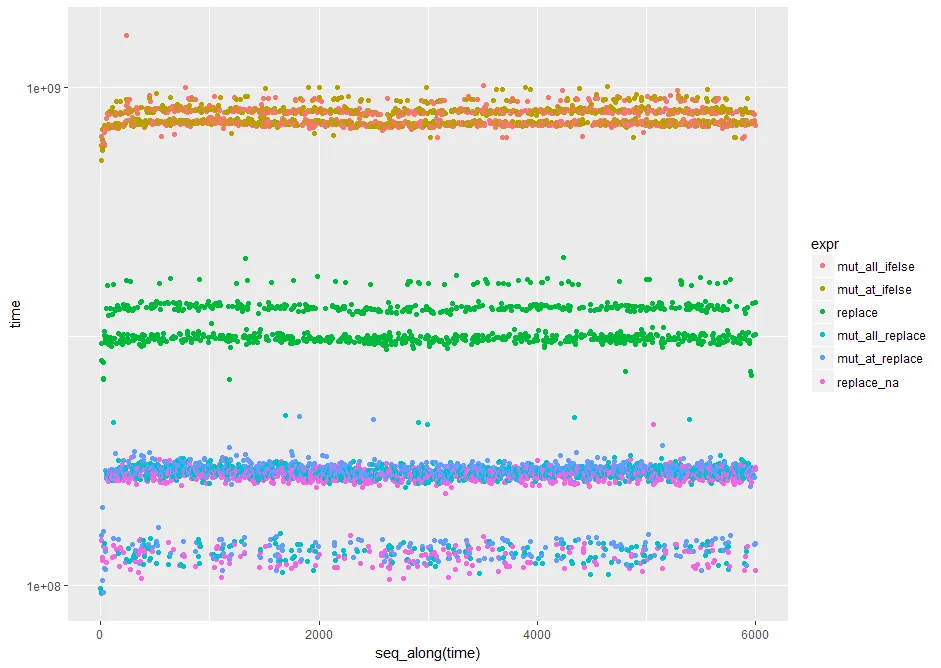
library(ggplot2) #nice log plot of the output
qplot(y=time, data=op, colour=expr) + scale_y_log10()
为了测试子集赋值运算符,我最初运行了这些测试。
set.seed(24)
> Book1 <- as.data.frame(matrix(sample(c(NA, 1:5), 1e8 *12, replace=TRUE),
+ dimnames = list(NULL, paste0("var", 1:12)), ncol=12))
> system.time({
+ Book1 %>% mutate_all(funs(ifelse(is.na(.), 0, .))) })
user system elapsed
52.79 24.66 77.45
>
> system.time({
+ Book1 %>% mutate_at(funs(ifelse(is.na(.), 0, .)), .cols = c(1:12)) })
user system elapsed
52.74 25.16 77.91
>
> system.time({
+ Book1[is.na(Book1)] <- 0 })
user system elapsed
16.65 7.86 24.51
>
> system.time({
+ Book1 %>% replace_na(list(var1 = 0, var2 = 0, var3 = 0, var4 = 0, var5 = 0, var6 = 0, var7 = 0, var8 = 0, var9 = 0,var10 = 0, var11 = 0, var12 = 0)) })
user system elapsed
3.54 2.13 5.68
>
> system.time({
+ Book1 %>% mutate_at(funs(replace(., is.na(.), 0)), .cols = c(1:12)) })
user system elapsed
3.37 2.26 5.63
>
> system.time({
+ Book1 %>% mutate_all(funs(replace(., is.na(.), 0))) })
user system elapsed
3.33 2.26 5.58
>
> system.time({
+ Book1 %>% replace(., is.na(.), 0) })
user system elapsed
3.42 1.09 4.51
在这些测试中,基本的
replace()首先出现。在基准测试中,replace()排名较低,而tidyr的replace_na()微弱获胜。
重复运行单个测试,并在不同形状和大小的数据框上运行,始终发现基本的replace()领先。问题2:为什么它的基准性能是唯一一个与简单测试结果如此不符的结果?
更加令人困惑的是 -
问题3:为什么所有的
mutate_all/_at(replace())都比简单的replace()运行得快呢?
许多人报告了这个问题:http://datascience.la/dplyr-and-a-very-basic-benchmark/(以及那篇文章中的所有链接),但我还没有找到一个解释为什么会这样的原因,除了 hashing 和 C++ 的使用)特别感谢 Tyler Rinker:https://www.r-bloggers.com/microbenchmarking-with-r/ 和 akrun:https://dev59.com/n57ha4cB1Zd3GeqPcwAp#41530071
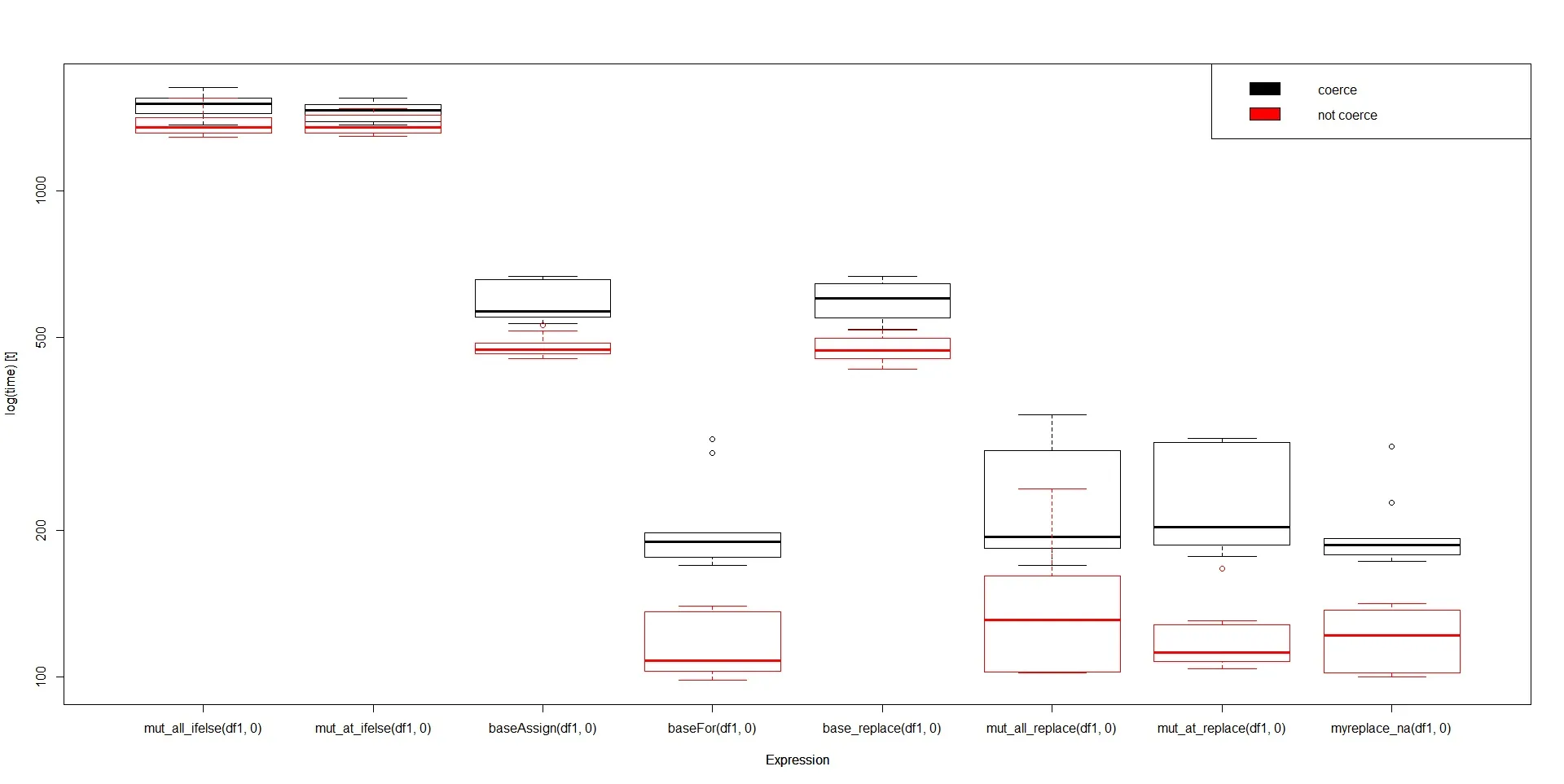
{}包装它 --{ df1[is.na(df1)] <- 0 }。顺便提一下,注意df1和Book1都是“整数”,在所有情况下你都在强制转换为“数字”。将0替换为0L应该可以提高速度。此外,在基准测试时,请注意Book1[is.na(Book1)] <- 0将实际的Book1替换为从“整数”到“数字”的强制转换的Book1,而所有后续情况都具有不需要强制转换的优势。为了避免强制转换原始数据,请使用函数或local进行包装。最后,我认为一个有效的方法是for(j in 1:ncol(df1)) df1[[j]][is.na(df1[[j]])] = 0L。 - alexis_lazfor循环是最快的替代方案之一,因为它只执行必须完成的最小操作来替换向量中的值。在for循环中发生的所有子集都只使用原始函数而不是“data.frame”方法进行[和[<-,这包括了显著的开销。唯一能够“击败”(但并不显著)循环内的这种一系列操作的事情就是就地修改;这是基本R不支持的。 - alexis_lazdf1 [[1]] [is.na(df1 [[1]])] = 0L; df1 [[2]] [is.na(df1 [[2]])] = 0L; etc ...但包装为方便且合理的代码在“for”循环中。无论是用户代码、R函数还是内部代码,都必须对“data.frame”列向量进行迭代选择。 - alexis_laz