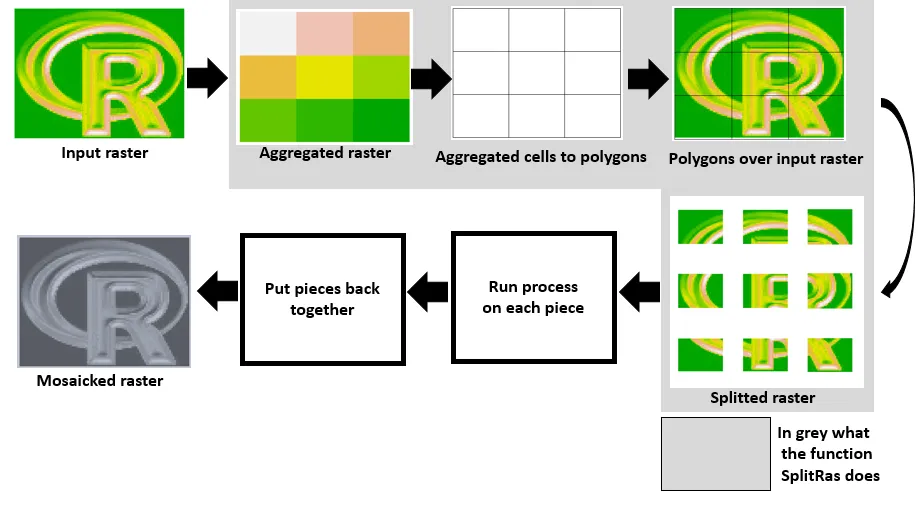
这里是使用“raster”包的另一种方法。该函数将栅格空间聚合为要切割的大小,聚合后的栅格单元被转换为多边形,然后使用每个多边形的范围来裁剪输入栅格。
我相信有更复杂和紧凑的方法来做到这一点,但这种方法对我很有效,而且我也觉得很直观。希望您也会发现它有用。请注意,下面的第4和第5部分仅用于测试,不是函数的一部分。
第一部分:加载并绘制示例栅格数据
logo <- raster(system.file("external/rlogo.grd", package="raster"))
plot(logo,axes=F,legend=F,bty="n",box=FALSE)
第二部分:函数本身:
SplitRas <- function(raster,ppside,save,plot){
h <- ceiling(ncol(raster)/ppside)
v <- ceiling(nrow(raster)/ppside)
agg <- aggregate(raster,fact=c(h,v))
agg[] <- 1:ncell(agg)
agg_poly <- rasterToPolygons(agg)
names(agg_poly) <- "polis"
r_list <- list()
for(i in 1:ncell(agg)){
e1 <- extent(agg_poly[agg_poly$polis==i,])
r_list[[i]] <- crop(raster,e1)
}
if(save==T){
for(i in 1:length(r_list)){
writeRaster(r_list[[i]],filename=paste("SplitRas",i,sep=""),
format="GTiff",datatype="FLT4S",overwrite=TRUE)
}
}
if(plot==T){
par(mfrow=c(ppside,ppside))
for(i in 1:length(r_list)){
plot(r_list[[i]],axes=F,legend=F,bty="n",box=FALSE)
}
}
return(r_list)
}
第三部分:测试函数
SplitRas(raster=logo,ppside=3,save=TRUE,plot=TRUE)
第四部分:对每个部分运行代码并将它们保存回目录中。
list2 <- list()
for(i in 1:9){
rx <- raster(paste("SplitRas",i,".tif",sep=""))
writeRaster(piece_processed,filename=paste("SplitRas",i,sep=""),
format="GTiff",datatype="FLT4S",overwrite=TRUE)
}
第五部分:让我们把碎片拼回一起。
list2 <- list()
for(i in 1:9){
rx <- raster(paste("SplitRas",i,".tif",sep=""))
list2[[i]] <- rx
}
list2$fun <- max
rast.mosaic <- do.call(mosaic,list2)
plot(rast.mosaic,axes=F,legend=F,bty="n",box=FALSE)
writeRaster(rast.mosaic,filename=paste("Mosaicked_ras",sep=""),
format="GTiff",datatype="FLT4S",overwrite=TRUE)

raster包专门用于 "地理数据分析和建模"。 - user3710546