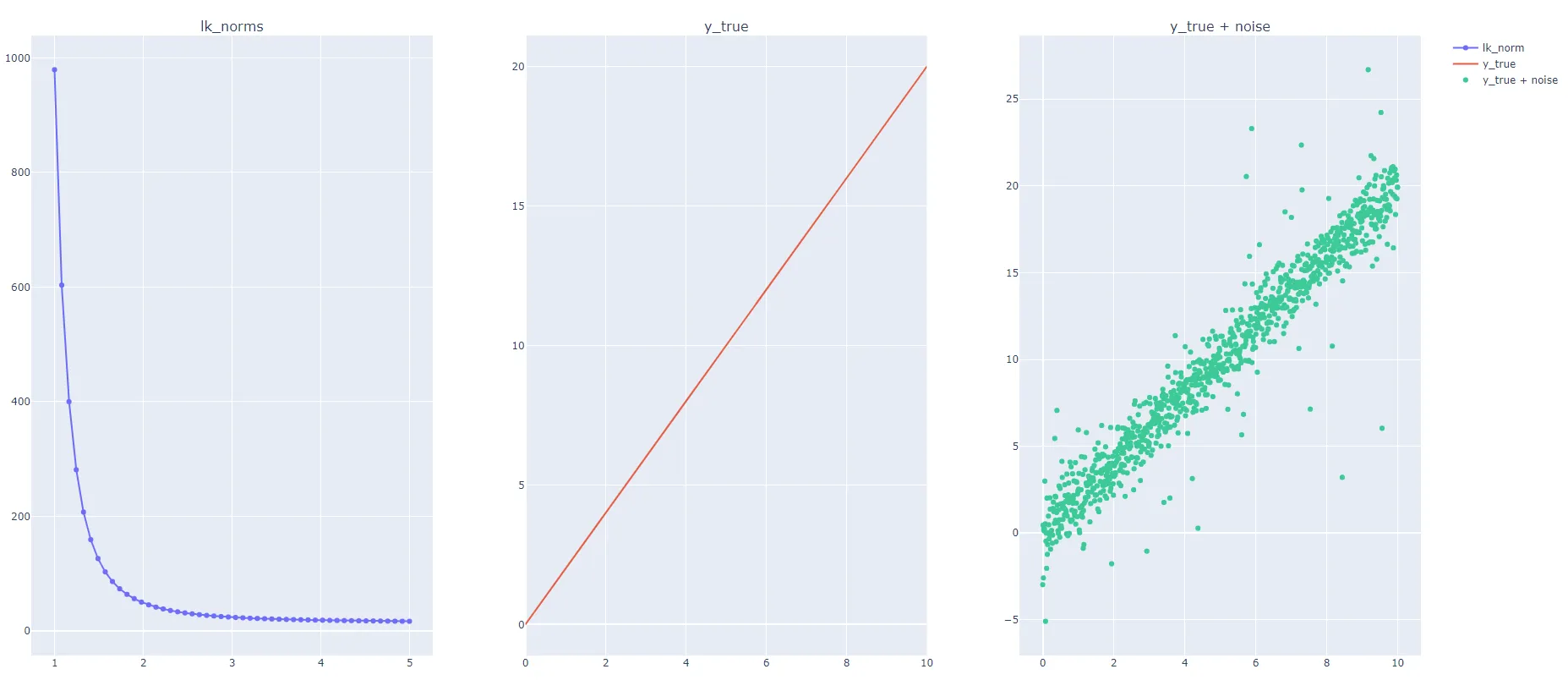
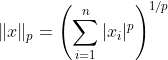( RMSE 和 MAE 都是衡量两个向量之间距离的方法:预测向量和目标值向量。有各种距离度量或范数可供选择。通常情况下,计算向量的大小或长度通常直接需要或作为更广泛的向量或向量矩阵运算的一部分。
尽管在回归任务中RMSE通常是首选的性能度量,但在某些情况下,您可能更喜欢使用另一个函数。例如,如果数据集中有许多异常值实例,则可以考虑使用平均绝对误差(MAE)。
更正式地说,范数指数越高,它就越关注大的值而忽略小的值。这就是为什么RMSE比MAE更敏感于异常值的原因。) 来源:《Python机器学习基础教程》
因此,在任何数据集中理想情况下,如果我们有很多异常值,则表示“表示预测和真实标签之间的绝对差异的向量的范数;类似于下面代码中的y_diff”应增长,如果我们增加范数...换句话说,RMSE应该大于MAE。--> 如果有误,请纠正 <--
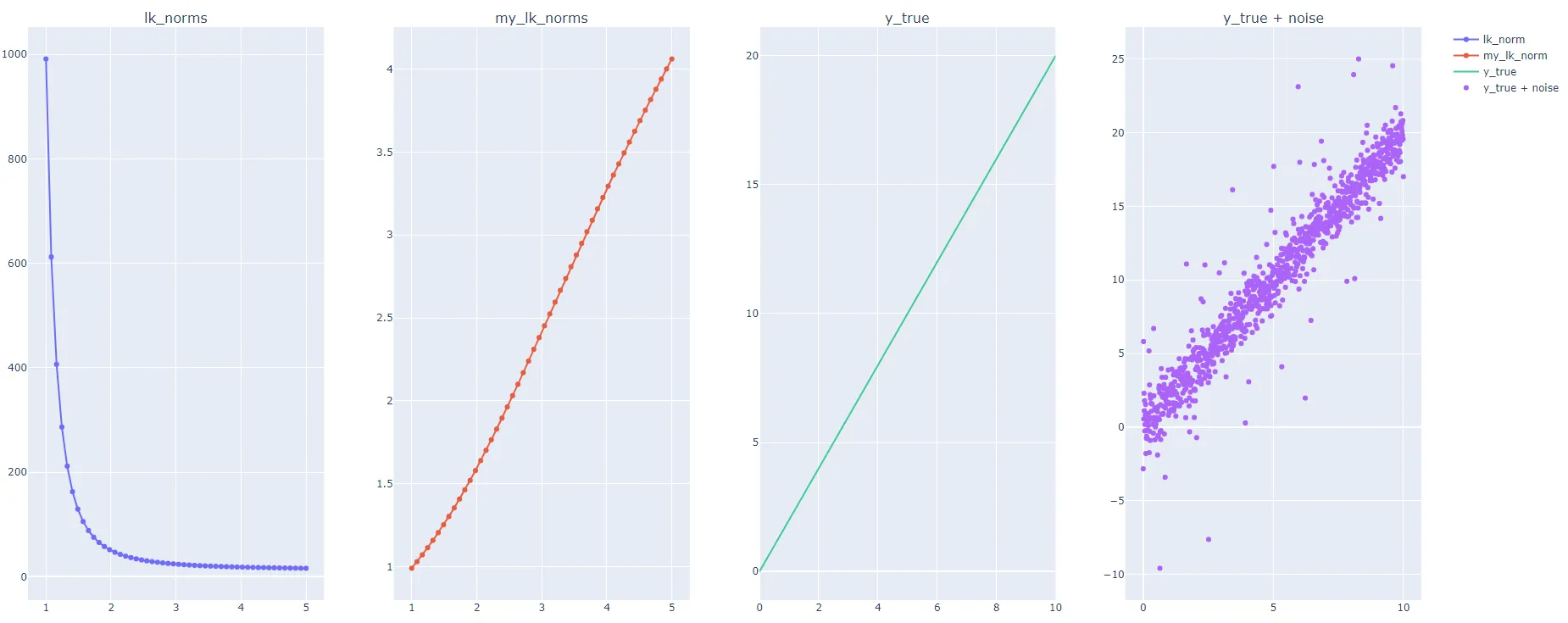
根据这个定义,我生成了一个随机数据集,并在其中添加了许多异常值,如下面的代码所示。我计算了残差或y_diff的lk_norm,对于许多k值(从1到5),但我发现当K值增加时,lk_norm会降低;然而,我期望RMSE,即norm=2,大于MAE,即norm=1。
我很想了解LK范数如何在增加K(即顺序)时降低,这与上面的定义相反。
感谢您提前提供的任何帮助!
代码:
import numpy as np
import plotly.offline as pyo
import plotly.graph_objs as go
from plotly import tools
num_points = 1000
num_outliers = 50
x = np.linspace(0, 10, num_points)
# places where to add outliers:
outlier_locs = np.random.choice(len(x), size=num_outliers, replace=False)
outlier_vals = np.random.normal(loc=1, scale=5, size=num_outliers)
y_true = 2 * x
y_pred = 2 * x + np.random.normal(size=num_points)
y_pred[outlier_locs] += outlier_vals
y_diff = y_true - y_pred
losses_given_lk = []
norms = np.linspace(1, 5, 50)
for k in norms:
losses_given_lk.append(np.linalg.norm(y_diff, k))
trace_1 = go.Scatter(x=norms,
y=losses_given_lk,
mode="markers+lines",
name="lk_norm")
trace_2 = go.Scatter(x=x,
y=y_true,
mode="lines",
name="y_true")
trace_3 = go.Scatter(x=x,
y=y_pred,
mode="markers",
name="y_true + noise")
fig = tools.make_subplots(rows=1, cols=3, subplot_titles=("lk_norms", "y_true", "y_true + noise"))
fig.append_trace(trace_1, 1, 1)
fig.append_trace(trace_2, 1, 2)
fig.append_trace(trace_3, 1, 3)
pyo.plot(fig, filename="lk_norms.html")
输出:
最后,我想知道什么情况下使用L3或L4范数等等...