寻找R中距离所有其他点最远的点
10
- Adam
7
1我不确定这个问题的规范是否恰当。你使用的是哪个函数来聚合从特定点到其他点的距离? - lmo
你对REST的定义是什么?因为本质上,你正在尝试找到在整个数据集中是异常值的观察结果。通常,人们会通过找到只在单个列(变量/特征)方面更远的观察结果来做到这一点,但你正在尝试与整个“blob”进行比较,明白吗? - Guilherme Marthe
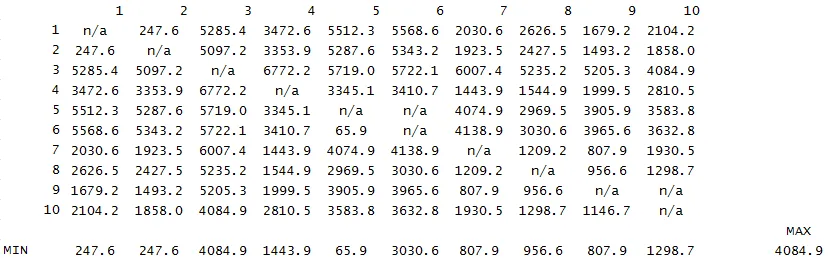
抱歉如果问题不够清晰 - 我在原帖中添加了一张图片,希望能展示我想要做的事情。谢谢您的帮助。 - Adam
1通过对数据进行查询,我可以查找每个点与任何其他点之间的最小距离,然后查找所有最小值中的最大距离。 - Adam
@amonk 我可能误解了问题,还是...?因为我看到的答案完全不同... - amonk
显示剩余2条评论
6个回答
4
从这个数据集开始:
现在,
这将为您提供到最近点的最大距离。如果您想知道这些点是哪些,可以进行额外步骤:
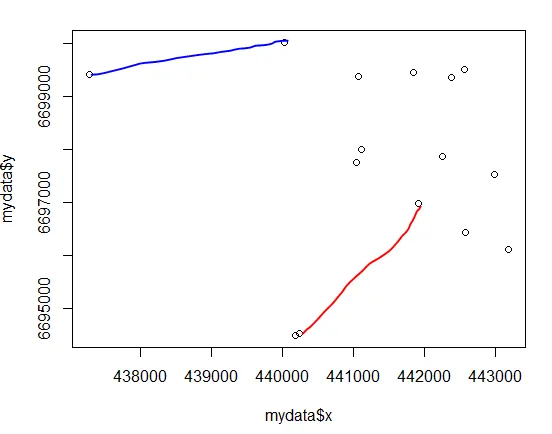
因此,点130和59是满足您条件的两个点。绘制如下图所示:
请注意,您会得到这些信息两次,因为两点之间的欧几里得距离是对称的(A -> B 和 B -> A 的长度相同)。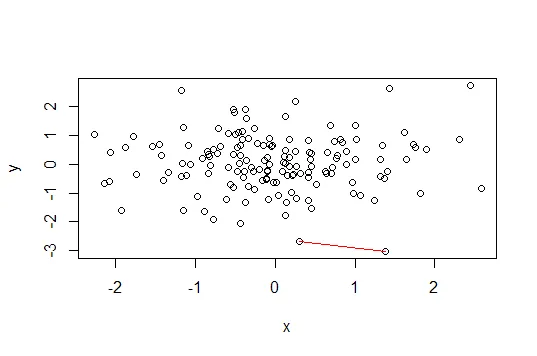
set.seed(100)
x <- rnorm(150)
y <- rnorm(150)
coord <- cbind(x,y)
dobj <- dist(coord)
现在,
dobj 是一个距离对象,但是你不能直接检查它。你需要先将其转换为矩阵,并确保不考虑点与自身之间的零距离:dmat <- as.matrix(dobj)
diag(dmat) <- NA
后一行将距离矩阵中的对角线值替换为NA。
现在您可以使用amonk的解决方案:
dmax <- max(apply(dmat,2,min,na.rm=TRUE))
这将为您提供到最近点的最大距离。如果您想知道这些点是哪些,可以进行额外步骤:
which(dmat == dmax, arr.ind = TRUE)
# row col
# 130 130 59
# 59 59 130
因此,点130和59是满足您条件的两个点。绘制如下图所示:
id <- which(dmat == dmax, arr.ind = TRUE)
plot(coord)
lines(coord[id[1,],], col = 'red')
请注意,您会得到这些信息两次,因为两点之间的欧几里得距离是对称的(A -> B 和 B -> A 的长度相同)。

- Joris Meys
1
如果你的初始数据框是df,你可以执行以下操作:
df<-NULL#initialize object
for(i in 1:10)#create 10 vectors with 10 pseudorandom numbers each
df<-cbind(df,runif(10))#fill the dataframe
cordf<-cor(df);diag(cordf)<-NA #create correlation matrix and set diagonal values to NA
因此:
[,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9] [,10]
[1,] NA -0.03540916 -0.29183703 0.49358124 0.79846794 0.29490246 0.47661166 -0.51181482 -0.04116772 -0.10797632
[2,] -0.03540916 NA 0.47550478 -0.24284088 -0.01898357 -0.67102287 -0.46488410 0.01125144 0.13355919 0.08738474
[3,] -0.29183703 0.47550478 NA -0.05203104 -0.26311149 0.01120055 -0.16521411 0.49215496 0.40571893 0.30595246
[4,] 0.49358124 -0.24284088 -0.05203104 NA 0.60558581 0.53848638 0.80623397 -0.49950396 -0.01080598 0.41798727
[5,] 0.79846794 -0.01898357 -0.26311149 0.60558581 NA 0.33295170 0.53675545 -0.54756131 0.09225002 -0.01925587
[6,] 0.29490246 -0.67102287 0.01120055 0.53848638 0.33295170 NA 0.72936185 0.09463988 0.14607018 0.19487579
[7,] 0.47661166 -0.46488410 -0.16521411 0.80623397 0.53675545 0.72936185 NA -0.46348644 -0.05275132 0.47619940
[8,] -0.51181482 0.01125144 0.49215496 -0.49950396 -0.54756131 0.09463988 -0.46348644 NA 0.64924510 0.06783324
[9,] -0.04116772 0.13355919 0.40571893 -0.01080598 0.09225002 0.14607018 -0.05275132 0.64924510 NA 0.44698207
[10,] -0.10797632 0.08738474 0.30595246 0.41798727 -0.01925587 0.19487579 0.47619940 0.06783324 0.44698207 NA
最终通过执行以下命令:
max(apply(cordf,2,min,na.rm=TRUE),na.rm = TRUE)#avoiding NA's
一个可以得到:
[1] -0.05275132
最大值是“局部”最小值。
编辑:
为了获取矩阵的索引>which(cordf==max(apply(cordf,2,min,na.rm=TRUE),na.rm = TRUE))
[1]68 77
或者为了获得坐标:
> which(cordf==max(apply(cordf,2,min,na.rm=TRUE),na.rm = TRUE), arr.ind = TRUE)
row col
[1,] 8 7
[2,] 7 8
- amonk
1
1请用
TRUE代替T。在控制台中输入T <- FALSE,然后再次运行代码以查看原因。 - Joris Meys1
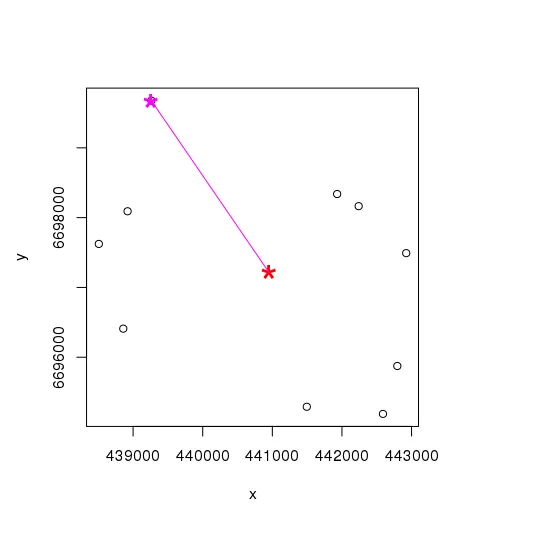
在我看来,你的空间点是以某种投影方式表示的。有人可能会说,与其他点最远的那个点,就是距离中心点(平均坐标)最远的点:
library(raster)
set.seed(21)
# create fake points
coords <- data.frame(x=sample(438000:443000,10),y=sample(6695000:6700000,10))
# calculate center
center <- matrix(colMeans(coords),ncol=2)
# red = center, magenta = furthest point (Nr.2)
plot(coords)
# furthest point #2
ix <- which.max(pointDistance(coords,center,lonlat = F))
points(center,col='red',pch='*',cex=3)
points(coords[ix,],col='magenta',pch='*',cex=3)
segments(coords[ix,1],coords[ix,2],center[1,1],center[1,2],col='magenta')
- Val
1
1这在数学上是不正确的。并不是因为一个点距离中心最远,那么从该点到另一个点的最小距离就是两个点之间的最大最小距离。在我的示例上尝试您的代码,您会发现选择了错误的点。 - Joris Meys
1
要找到与其他点最远的点,您可以这样做。我选择了中位距离,因为您说了点与其他数据最远。如果有一组非常接近的点,则中位数应该对此保持稳健。
可能也有一种使用分层聚类的方法,但目前我无法想起来。
可能也有一种使用分层聚类的方法,但目前我无法想起来。
set.seed(1234)
mat <- rbind(matrix(rnorm(100), ncol=2), c(-5,5), c(-5.25,4.75))
d <- dist(mat)
sort(apply(as.matrix(d), 1, median), decreasing = T)[1:5]
# 51 52 20 12 4
# 6.828322 6.797696 3.264315 2.806263 2.470919
- emilliman5
1
我写了一个方便的小函数,可以用来选择最大的行距。你可以使用n参数指定你想要最大的、第二大的等等。
getBigSegment <- function(x, y, n = 1){
a <- cbind(x,y)
d <- as.matrix(dist(a, method = "euclidean"))
sorted <- order(d, decreasing = T)
sub <- (1:length(d))[as.logical(1:length(sorted) %% 2)]
s <- which(d == d[sorted[sub][n]], arr.ind = T)
t(cbind(a[s[1],], a[s[2],]))
}
有一些类似于您自己的示例数据,您可以看到:
set.seed(100)
mydata <- data.frame(x = runif(10, 438000, 445000) + rpois(10, 440000),
y = runif(10, 6695000, 6699000) + rpois(10, 6996000))
# The function
getBigSegment(mydata$x, mydata$y)
# x y
#[1,] 883552.8 13699108
#[2,] 881338.8 13688458
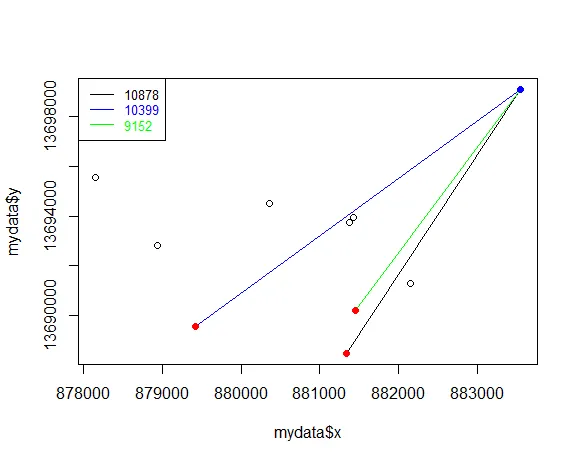
以下是我如何使用这样一个函数的可视化演示。
# easy plotting function
pointsegments <- function(z, ...) {
segments(z[1,1], z[1,2], z[2,1], z[2,2], ...)
points(z, pch = 16, col = c("blue", "red"))
}
plot(mydata$x, mydata$y) # points
top3 <- lapply(1:3, getBigSegment, x = mydata$x, y = mydata$y) # top3 longest lines
mycolors <- c("black","blue","green") # 3 colors
for(i in 1:3) pointsegments(top3[[i]], col = mycolors[i]) # plot lines
legend("topleft", legend = round(unlist(lapply(top3, dist))), lty = 1,
col = mycolors, text.col = mycolors, cex = .8) # legend
- Evan Friedland
1
这种方法首先使用
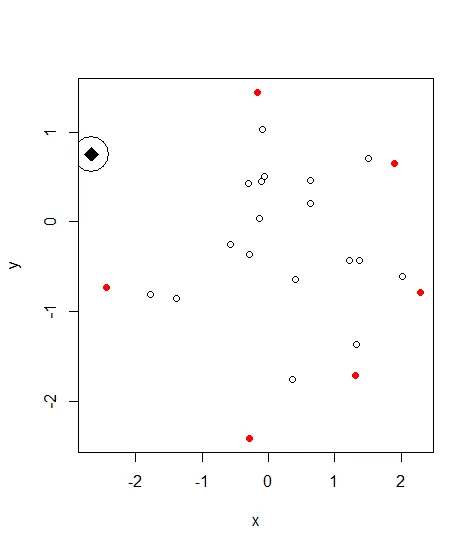
chull 来确定 extreme_points,即位于给定点的边界上的点。然后,对于每个 extreme_points,它通过排除该特定的 extreme_points 来计算 extreme_points 的 centroid。然后,它从 extreme_points 中选择离 centroid 最远的点。foo = function(X = all_points){
plot(X)
chull_inds = chull(X)
extreme_points = X[chull_inds,]
points(extreme_points, pch = 19, col = "red")
centroid = t(sapply(1:NROW(extreme_points), function(i)
c(mean(extreme_points[-i,1]), mean(extreme_points[-i,2]))))
distances = sapply(1:NROW(extreme_points), function(i)
dist(rbind(extreme_points[i,], centroid[i,])))
points(extreme_points[which.max(distances),], pch = 18, cex = 2)
points(X[chull_inds[which.max(distances)],], cex = 5)
return(X[chull_inds[which.max(distances)],])
}
set.seed(42)
all_points = data.frame(x = rnorm(25), y = rnorm(25))
foo(X = all_points)
# x y
#18 -2.656455 0.7581632
- d.b
网页内容由stack overflow 提供, 点击上面的可以查看英文原文,
原文链接
原文链接