我试图运行一个相对简单的glmer模型,但收到了Singular警告,我无法弄清原因。
在我的数据集中,有40个参与者参加了108次试验。他们回答了一个问题(回答被编码为正确/不正确 - 0/1),并在0到1的连续尺度上评估了他们的回答信心。
library(lme4)
library(tidybayes)
library(tidyverse)
set.seed(5)
n_trials = 108
n_subjs = 40
data =
tibble(
subject = as.factor(rep(c(1:n_subjs), n_trials)),
correct = sample(c(0,1), replace=TRUE, size=(n_trials*n_subjs)),
confidence = runif(n_trials*n_subjs)
)
我希望运行一个混合效应的逻辑回归,以估计每个参与者仅将高置信度与正确响应相关联的能力。这意味着,我有充分的理由在模型中添加置信度的随机斜率。
我感兴趣的最简单模型是:
model = glmer(correct ~ confidence + (confidence|subject) ,
data = data,
family = binomial)
边界(单数)契合:参见?isSingular,以及
> isSingular(model)
[1] TRUE
所以我将模型简化到超出有用的程度,得到了同样的问题:
model = glmer(correct ~ confidence + (1|subject) ,
data = data,
family = binomial)
我尝试对置信度进行分组(我相信还有更优雅的方法),但这并没有帮助:
#Initialize as vector of 0s
data$confidence_binned <- numeric(dim(data)[1])
nbins = 4
bins=seq(0,1,length.out = (nbins+1))
for (b in 1:(length(bins)-1)) {
data$confidence_binned[data$confidence>=bins[b] & data$confidence<bins[b+1]] = b
}
data$confidence_binned[data$confidence_binned==1]=nbins
model = glmer(correct ~ confidence_binned + (confidence_binned|subject) ,
data = data,
family = binomial)
boundary (singular) fit: 参见 ?isSingular。
关于 isSingular 警告有很多帖子和 SO 问题,但我找到的所有内容都说模型对数据太复杂,解决方案通常是“保持最大化”。然而,这个模型尽可能简单,我很困惑,即使进行了足够的试验,它仍然失败(听起来像是)。
我还尝试更改控制器,但没有帮助:
ctrl = glmerControl(optimizer = "bobyqa",
boundary.tol = 1e-5,
calc.derivs=TRUE,
use.last.params=FALSE,
sparseX = FALSE,
tolPwrss=1e-7,
compDev=TRUE,
nAGQ0initStep=TRUE,
## optimizer args
optCtrl = list(maxfun = 1e5))
model <- glmer(correct ~ confidence_binned + (confidence_binned|subject),
data=data,
verbose=T,
control=ctrl,
family = binomial)
任何有关数据注意事项的帮助或指针都将不胜感激。
回应评论的编辑:
ggplot(data,aes(x=subject, y=correct)) + stat_summary(fun.data=mean_cl_normal)的结果如下图所示:
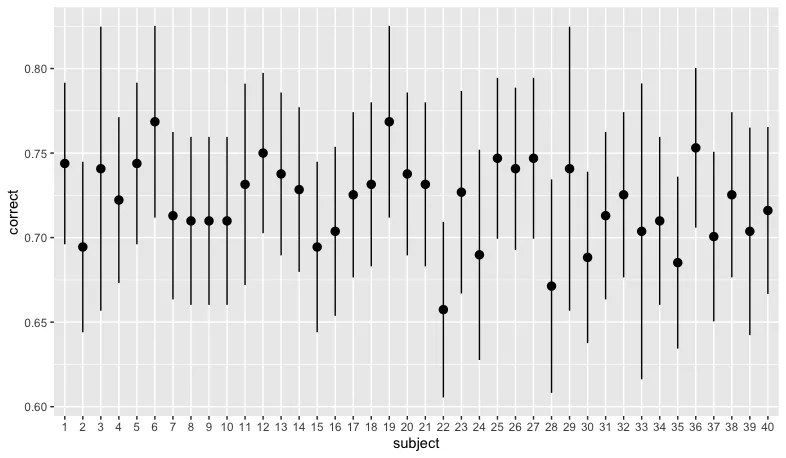
ggplot(data,aes(x = subject,y = correct))+ stat_summary(fun.data = mean_cl_normal)看起来如何? - user20650