我可以通过以下代码在scikit中进行PCA: X_train有279180行和104列。
from sklearn.decomposition import PCA
pca = PCA(n_components=30)
X_train_pca = pca.fit_transform(X_train)
现在,当我想要将特征向量投影到特征空间时,我必须执行以下操作:
""" Projection """
comp = pca.components_ #30x104
com_tr = np.transpose(pca.components_) #104x30
proj = np.dot(X_train,com_tr) #279180x104 * 104x30 = 297180x30
但我在犹豫这一步,因为Scikit的文档说:
components_: array, [n_components, n_features]
在特征空间中的主要轴,代表数据中方差最大的方向。
对我来说,它似乎已经被投影了,但是当我检查源代码时,它只返回特征向量。
那么如何正确地进行投影呢?
我的最终目标是计算重建的均方误差。
""" Reconstruct """
recon = np.dot(proj,comp) #297180x30 * 30x104 = 279180x104
""" MSE Error """
print "MSE = %.6G" %(np.mean((X_train - recon)**2))
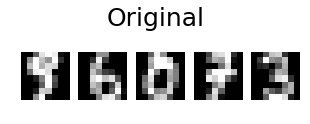
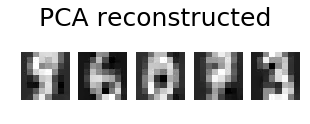
pca.fit_transform(X)与pca.fit(X).transform(X)给出相同的结果(它是一种优化的快捷方式)。其次,投影通常是从一个空间到同一空间的东西,因此在这里它将从信号空间到信号空间,具有应用两次就像应用一次的属性。在这里,它将是f = lambda X:pca.inverse_transform(pca.transform(X))。您可以检查f(f(X))== f(X)。因此,我会称之为投影。pca.transform正在获取载荷。最终只是术语问题。 - eickenbergassert_array_almost_equal(VT[:30], pca.components_)并不总是正确的。在PCA的实现中,U和V之间的符号被混合了。为了模仿这种混合,将U、S、VT = np.linalg.svd(Xtrain - Xtrain.mean(0))替换为U、S、VT = np.linalg.svd(Xtrain - Xtrain.mean(0), full_matrices=False),然后插入from sklearn.utils.extmath import svd_flip,接着是U、VT = svd_flip(U, VT)。 - Stanloss = ((X_train - X_projected) ** 2).mean()中的X_train是否替换了代码中之前定义的Xtrain变量? - Josmoor98