我在.csv文件中输入了一个包含Unicode符号如:\U00B5 g/dL的文本字符串。同时也可以在R数据框中读取.csv文件:
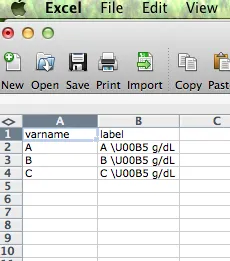
test=read.csv("test.csv")
\U00B5 将产生微符号- µ。R将其读入数据文件中(\U00B5)。但是,当我打印字符串时,它显示为 \\U00B5 g/dL。
另外,手动输入代码可以正常工作。
varname <- c("a", "b", "c")
labels <- c("A \U00B5 g/dL", "B \U00B5 g/dL", "C \U00B5 g/dL")
df <- data.frame(varname, labels)
test <- data.frame(varname, labels)
test
# varname labels
# 1 a A µ g/dL
# 2 b B µ g/dL
# 3 c C µ g/dL
我想知道如何在这种情况下去除转义符号\并打印出该符号。 或者,是否有另一种方法可以在R中打印该符号。
非常感谢您的帮助!
read.csv()函数调用中使用fileEncoding="UTF-8", allowEscapes=T。 - Alex A..csv文件导入,它会添加\\。 - outboundbird