感谢提供可重现的示例。这是相当基础的R语言知识。您可以将值分配给向量中的选定元素(请注意,数组具有维度,而您提供的是一个向量而不是数组):
> pred_precipitation[pred_precipitation<0] <- 0
> pred_precipitation
[1] 1.2091281 0.0000000 7.7665555 0.0000000 0.0000000 0.0000000 0.5151504 0.0000000 1.8281251
[10] 0.5098688 2.8370263 0.4895606 1.5152191 4.1740177 7.1527742 2.8992215 4.5322934 6.7180530
[19] 0.0000000 1.1914052 3.6152333 0.0000000 0.3778717 0.0000000 1.4940469
性能测试战争!
@James 发现了一种更快的方法,并在评论中留下了它。我为他点了赞,尽管我知道他的胜利将是短暂的。
首先,我尝试编译,但这似乎对任何人都没有帮助:
p <- rnorm(10000)
gsk3 <- function(x) { x[x<0] <- 0; x }
jmsigner <- function(x) ifelse(x<0, 0, x)
joshua <- function(x) pmin(x,0)
james <- function(x) (abs(x)+x)/2
library(compiler)
gsk3.c <- cmpfun(gsk3)
jmsigner.c <- cmpfun(jmsigner)
joshua.c <- cmpfun(joshua)
james.c <- cmpfun(james)
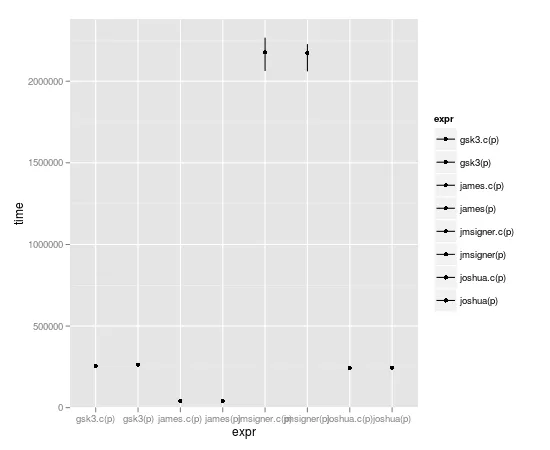
microbenchmark(joshua(p),joshua.c(p),gsk3(p),gsk3.c(p),jmsigner(p),james(p),jmsigner.c(p),james.c(p))
expr min lq median uq max
1 gsk3.c(p) 251.782 255.0515 266.8685 269.5205 457.998
2 gsk3(p) 256.262 261.6105 270.7340 281.3560 2940.486
3 james.c(p) 38.418 41.3770 43.3020 45.6160 132.342
4 james(p) 38.934 42.1965 43.5700 47.2085 4524.303
5 jmsigner.c(p) 2047.739 2145.9915 2198.6170 2291.8475 4879.418
6 jmsigner(p) 2047.502 2169.9555 2258.6225 2405.0730 5064.334
7 joshua.c(p) 237.008 244.3570 251.7375 265.2545 376.684
8 joshua(p) 237.545 244.8635 255.1690 271.9910 430.566
等等!Dirk写了这个Rcpp东西。一个完全不懂C++的人能否阅读他的JSS论文、调整他的例子并编写出所有函数中最快的函数呢?请关注,亲爱的听众。
library(inline)
cpp_if_src <- '
Rcpp::NumericVector xa(a);
int n_xa = xa.size();
for(int i=0; i < n_xa; i++) {
if(xa[i]<0) xa[i] = 0;
}
return xa;
'
cpp_if <- cxxfunction(signature(a="numeric"), cpp_if_src, plugin="Rcpp")
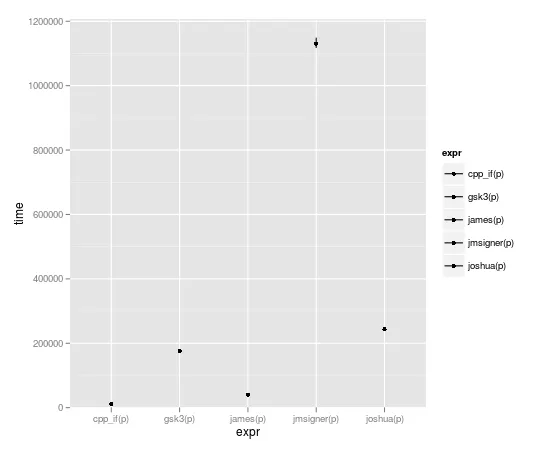
microbenchmark(joshua(p),joshua.c(p),gsk3(p),gsk3.c(p),jmsigner(p),james(p),jmsigner.c(p),james.c(p), cpp_if(p))
expr min lq median uq max
1 cpp_if(p) 8.233 10.4865 11.6000 12.4090 69.512
2 gsk3(p) 170.572 172.7975 175.0515 182.4035 2515.870
3 james(p) 37.074 39.6955 40.5720 42.1965 2396.758
4 jmsigner(p) 1110.313 1118.9445 1133.4725 1164.2305 65942.680
5 joshua(p) 237.135 240.1655 243.3990 250.3660 2597.429
是的,船长。
即使您没有对输入变量p进行赋值,该代码仍会修改它。如果您想避免这种行为,您需要克隆该变量:
cpp_ifclone_src <- '
Rcpp::NumericVector xa(Rcpp::clone(a));
int n_xa = xa.size();
for(int i=0; i < n_xa; i++) {
if(xa[i]<0) xa[i] = 0;
}
return xa;
'
cpp_ifclone <- cxxfunction(signature(a="numeric"), cpp_ifclone_src, plugin="Rcpp")
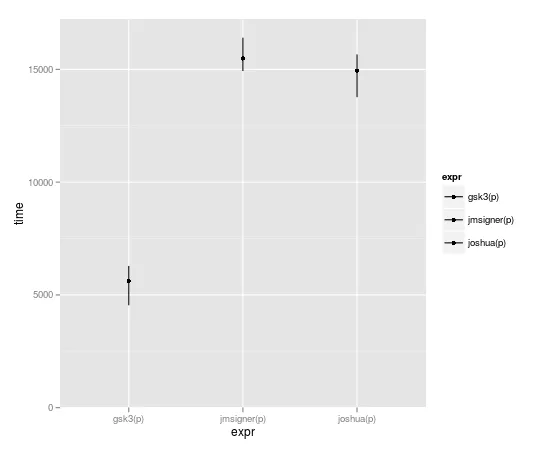
这不幸会削弱速度优势。


p而不进行赋值吗?当我尝试时似乎并没有。 - Aaron left Stack Overflow