使用以下示例数据,为这个非常有趣的问题提供一个候选解决方案。为了适应受罚VAR,我使用了最近发布的
BigVAR包。此时它不具备产生FEVDs、历史分解、预测范围图等通常的额外功能,但通过
cv.BigVAR可以获得来自约简形式模型的所有必要输出。这是我下面的第一步所做的。我将把调整约简形式估计的工作留给你,使用软件包功能进行微调。
library(BigVAR)
library(expm)
library(data.table)
data(Y)
p = 4
k = ncol(Y)
N = nrow(Y) - p
mod1 = constructModel(Y,p,"Basic",gran=c(150,10),RVAR=FALSE,h=1,cv="Rolling",MN=FALSE,verbose=FALSE,IC=TRUE)
results = cv.BigVAR(mod1)
A = results@betaPred[,2:ncol(results@betaPred)]
Sigma = crossprod(resid(varres))/(N-(k*p)-1)
从那里,我们可以使用标准公式计算FEVD。为了介绍方便,我强烈推荐Lutz Kilian和Helmut Lütkepohl的《结构向量自回归分析》第4章
Chapter 4。下面实现了我们从简化形式IRFs到MSPE再到R中计算FEVD所需的一切公式。这可能需要花费很多时间,而且我没有时间来优化这段代码,因此可能还可以更有效地完成,但希望它仍然具有信息性。请注意,我使用标准的Choleski分解来识别VAR,因此变量排序的通常含义适用。
compute_Phi = function(p, k, A_comp, n.ahead) {
J = matrix(0,nrow=k,ncol=k*p)
diag(J) = 1
Phi = lapply(1:n.ahead, function(i) {
J %*% (A_comp %^% (i-1)) %*% t(J)
})
return(Phi)
}
compute_theta_kj_sq = function(Theta, n.ahead) {
theta_kj_sq = lapply(1:n.ahead, function(h) {
out = sapply(1:ncol(Theta[[h]]), function(k) {
terms_to_sum = lapply(1:h, function(i) {
Theta[[i]][k,]**2
})
theta_kj_sq_h = Reduce(`+`, terms_to_sum)
})
colnames(out) = colnames(Theta[[h]])
return(out)
})
return(theta_kj_sq)
}
compute_mspe = function(Theta, n.ahead=10) {
mspe = lapply(1:n.ahead, function(h) {
terms_to_sum = lapply(1:h, function(i) {
tcrossprod(Theta[[i]])
})
mspe_h = Reduce(`+`, terms_to_sum)
})
}
fevd = function(A, Sigma, n.ahead) {
k = dim(A)[1]
p = dim(A)[2]/k
if (p>1) {
A_comp = VarptoVar1MC(A,p,k)
} else {
A_comp = A
}
Phi = compute_Phi(p,k,A_comp,n.ahead)
P = t(chol.default(Sigma))
B_0 = solve(P)
Theta = lapply(1:length(Phi), function(i) {
Phi[[i]] %*% solve(B_0)
})
theta_kj_sq = compute_theta_kj_sq(Theta, n.ahead)
mspe = compute_mspe(Theta, n.ahead)
fevd_list = lapply(1:k, function(k) {
t(sapply(1:length(mspe), function(h) {
mspe_k = mspe[[h]][k,k]
theta_k_sq = theta_kj_sq[[h]][,k]
fevd = theta_k_sq/mspe_k
}))
})
fevd_tidy = data.table::rbindlist(
lapply(1:length(fevd_list), function(k) {
fevd_k = data.table::melt(data.table::data.table(fevd_list[[k]])[,h:=.I], id.vars = "h", variable.name = "j")
fevd_k[,k:=paste0("V",k)]
data.table::setcolorder(fevd_k, c("k", "j", "h"))
})
)
return(fevd_tidy)
}
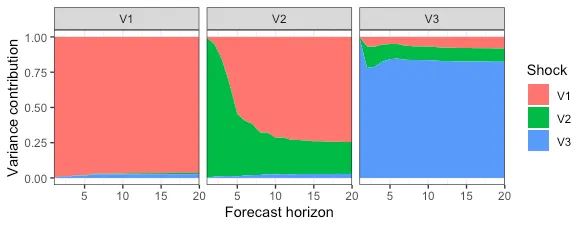
最后,让我们实现
n.ahead=20 的公式并绘制结果。
fevd_res = fevd(A, Sigma, 20)
library(ggplot2)
p = ggplot(data=fevd_res) +
geom_area(aes(x=h, y=value, fill=j)) +
facet_wrap(k ~ .) +
scale_x_continuous(
expand=c(0,0)
) +
scale_fill_discrete(
name = "Shock"
) +
labs(
y = "Variance contribution",
x = "Forecast horizon"
) +
theme_bw()
p
希望这对你有所帮助,如果有任何后续问题,请随时提出。
最后要注意的一点是:我已经使用你在问题中提到的
vars包将FEVD函数与标准VAR的结果进行了比较,并且检查通过(如下所示)。但这是我的“单元测试”范围。代码尚未经过任何人的审核,请自行查阅公式。如果您或其他人发现任何错误,我将非常感谢您的反馈。
编辑1
为了完整起见,添加了一个快速比较
vars::fevd返回的结果和上述自定义函数的结果。
library(vars)
p = 4
k = ncol(Y)
N = nrow(Y) - p
colnames(Y) = sprintf("V%i", 1:ncol(Y))
n.ahead = 20
varres = vars::VAR(Y,p)
Sigma = crossprod(resid(varres))/(N-(k*p)-1)
A = t(
sapply(coef(varres), function(i) {
i[,1]
})
)
A = A[,1:(ncol(A)-1)]
fevd_pkg = vars::fevd(varres, n.ahead)
fevd_cus = fevd(A, Sigma, n.ahead)
现在比较第一个变量的输出:
>
> head(fevd_pkg$V1)
V1 V2 V3
[1,] 1.0000000 0.00000000 0.00000000
[2,] 0.9399842 0.01303013 0.04698572
[3,] 0.9422918 0.01062750 0.04708065
[4,] 0.9231440 0.01409313 0.06276291
[5,] 0.9305901 0.01335727 0.05605267
[6,] 0.9093144 0.01278727 0.07789833
>
> head(dcast(fevd_cus[k=="V1"], k+h~j, value.var = "value"))
k h V1 V2 V3
1: V1 1 1.0000000 0.00000000 0.00000000
2: V1 2 0.9399842 0.01303013 0.04698572
3: V1 3 0.9422918 0.01062750 0.04708065
4: V1 4 0.9231440 0.01409313 0.06276291
5: V1 5 0.9305901 0.01335727 0.05605267
6: V1 6 0.9093144 0.01278727 0.07789833
编辑2
如果不依赖于vars :: VAR()的输出,您可以使用以下函数在R中获得广义FEVD。我已经回收了frequencyConnectedness :: genFEVD的一些源代码。
library(frequencyConnectedness)
genFEVD_cus = function(
A,
Sigma,
n.ahead,
no.corr=F
) {
k = dim(A)[1]
p = dim(A)[2]/k
if (p>1) {
A_comp = BigVAR::VarptoVar1MC(A,p,k)
} else {
A_comp = A
}
if (no.corr) {
Sigma = diag(diag(Sigma))
}
Phi = compute_Phi(p,k,A_comp,n.ahead+1)
denom = diag(Reduce("+", lapply(Phi, function(i) i %*% Sigma %*%
t(i))))
enum = Reduce("+", lapply(Phi, function(i) (i %*% Sigma)^2))
tab = sapply(1:nrow(enum), function(j) enum[j, ]/(denom[j] *
diag(Sigma)))
tab = t(apply(tab, 2, function(i) i/sum(i)))
return(tab)
}
继续上面的例子(Edit 1),将自定义函数的结果与依赖于vars::VAR()输出的frequencyConnectedness::genFEVD的结果进行比较:
> frequencyConnectedness::genFEVD(varres, n.ahead)
V1 V2 V3
[1,] 0.89215734 0.02281892 0.08502374
[2,] 0.72025050 0.19335481 0.08639469
[3,] 0.08328267 0.11769438 0.79902295
> genFEVD_cus(A, Sigma, n.ahead)
V1 V2 V3
[1,] 0.89215734 0.02281892 0.08502374
[2,] 0.72025050 0.19335481 0.08639469
[3,] 0.08328267 0.11769438 0.79902295
