这里是我用zoo编写的一些代码-我没有使用过xts,所以不知道是否可以应用相同的函数。希望有所帮助!
函数
以下函数计算原始数据每个时间间隔与给定时间间隔重叠的分数(注意:在以下所有代码中,变量名ta1和ta2指代给定时间间隔的开始和结束(例如需要作为输出的每个等间隔),而tb1和tb2则指代原始数据的(不等)时间间隔的开始和结束):
frac.overlap <- function(ta1,ta2,tb1,tb2){
if(tb1 <= ta1 & tb2 >= ta2) {
frac <- as.numeric(difftime(ta2,ta1,units="secs"))/as.numeric(difftime(tb2,tb1,units="secs"))
} else if(tb1 >= ta1 & tb2 <= ta2) {
frac <- 1
} else if(tb1 <= ta1 & tb2 >= ta1) {
frac <- as.numeric(difftime(tb2,ta1,units="secs"))/as.numeric(difftime(tb2,tb1,units="secs"))
} else if (tb1 <= ta2 & tb2 >= ta2){
frac <- as.numeric(difftime(ta2,tb1,units="secs"))/as.numeric(difftime(tb2,tb1,units="secs"))
} else {
frac <- 0
}
return(frac)
}
下一个函数确定原始数据集中与当前考虑的区间
ta1-
ta2 重叠的记录:
check.overlap <- function(ta1,ta2,tb1,tb2){
ov <- vector("logical",4)
ov[1] <- (tb1 <= ta1 & tb2 >= ta2) # Interval 2 starts earlier and ends later than interval 1
ov[2] <- (tb1 >= ta1 & tb2 <= ta2) # Interval 2 is fully contained within interval 1
ov[3] <- (tb1 <= ta1 & tb2 >= ta1) # Interval 2 partly overlaps with interval 1 (starts earlier, ends earlier)
ov[4] <- (tb1 <= ta2 & tb2 >= ta2) # Interval 2 partly overlaps with interval 1 (starts later, ends later)
return(as.logical(sum(ov))) # Gives TRUE if at least one element of ov is TRUE, otherwise FALSE
}
(注意:这在您提供的示例数据中效果很好,但在更大的数据集上,我发现它速度非常慢。由于我编写此代码以重新采样具有固定时间步长的时间序列,因此我通常使用固定间隔来完成此步骤,这样可以显著提高速度。很可能很容易修改代码(请参见下一个函数的代码),以根据原始数据的间隔加快此步骤。)
下一个函数使用前两个函数来计算间隔
ta1-
ta2的重新采样值:
fracres <- function(tstart,interval,input){
ta1 <- tstart
ta2 <- tstart + interval
ind <- index(input)
ind1 <- index(lag(input,-1))
recs <- which(sapply(1:length(ind),function(x) check.overlap(ta1,ta2,ind[x],ind1[x])))
if(length(recs) > 0){
fracs <- sapply(1:length(recs), function(x) frac.overlap(ta1,ta2,ind[recs[x]],ind1[recs[x]]))
return(sum(coredata(input)[recs]*fracs))
} else {
return(0)
}
}
(如果原始时间步长和新时间步长之间的最大时间差已知,则可以使用注释掉的行来获取相关记录。)
应用程序
首先,将您的示例数据作为zoo对象读入:
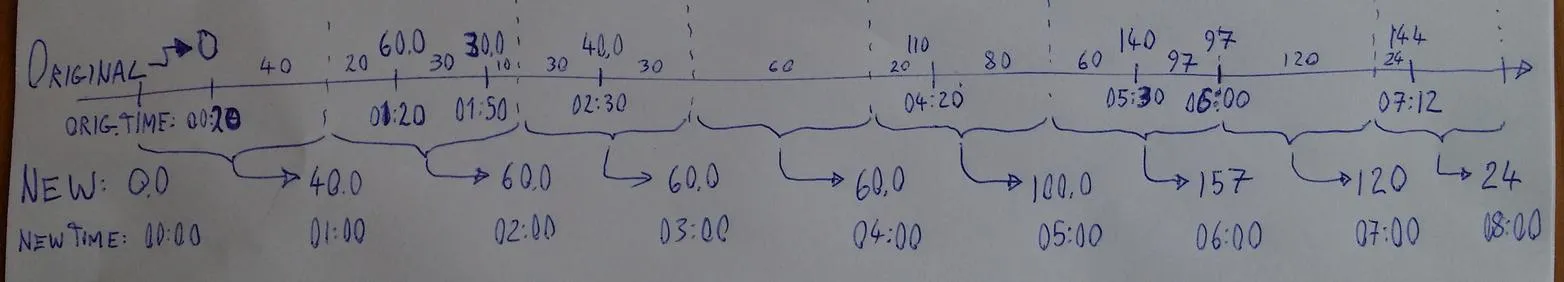
sample_zoo <- read.zoo(text='
2016-07-01 00:00:20, 0.0
2016-07-01 00:01:20, 60.0
2016-07-01 00:01:50, 30.0
2016-07-01 00:02:30, 40.0
2016-07-01 00:04:20, 110.0
2016-07-01 00:05:30, 140.0
2016-07-01 00:06:00, 97.0
2016-07-01 00:07:12, 144.0
2016-07-01 00:08:09, 0.0
', sep=',', index=1, tz='', format="%Y-%m-%d %H:%M:%S")
看起来你的数据集包含瞬时值(“在 01:20 时,x 的值为60”)。由于我编写的代码是针对累加值的,时间戳的含义不同(“从 01:20 开始的记录的值为60”)。为了纠正这个问题,需要对记录进行移位:
sample_zoo <- lag(sample_zoo,1)
然后,我们定义一个序列的
POSIXct对象,对应所需的分辨率:
time.out <- seq.POSIXt(from=as.POSIXct("2016-07-01"),to=(as.POSIXct("2016-07-01")+(60*9)),by="1 min")
我们可以接着使用上述描述的
fracres函数:
data.out <- sapply(1:length(time.out), function(x) fracres(tstart=time.out[x],interval=60,input=sample_zoo))
索引和数据被合并到一个名为
zoo 的对象中:
zoo.out <- read.zoo(data.frame(time.out,data.out))
最后,时间序列再次向相反的方向移动一步:
zoo.out <- lag(zoo.out,-1)
2016-07-01 00:01:00 2016-07-01 00:02:00 2016-07-01 00:03:00 2016-07-01 00:04:00 2016-07-01 00:05:00 2016-07-01 00:06:00 2016-07-01 00:07:00 2016-07-01 00:08:00 2016-07-01 00:09:00
40 60 60 60 100 157 120 24 0
 我希望的方法不仅限于创建1分钟时间步长系列,而是适用于任何固定时间步长。
我希望的方法不仅限于创建1分钟时间步长系列,而是适用于任何固定时间步长。