在data.table中给定任意列名列表,我想将这些列的内容连接为一个字符串,并存储到新列中。我需要动态生成表达式来进行拼接,因为需要拼接的列并不总是相同的。
我怀疑我使用 eval(parse(...)) 方法的方式可以用更优美的方式替换,但下面的方法是目前我能得到的最快的方法。
对于1000万行数据,在这个样例数据上运行时间大约为21.7秒(基本R函数paste0稍微慢一些--23.6秒)。我的实际数据有18-20列被连接,并且最多有1亿行,所以减速变得更加不切实际了。
有什么想法可以加速吗?
目前的方法
library(data.table)
library(stringi)
RowCount <- 1e7
DT <- data.table(x = "foo",
y = "bar",
a = sample.int(9, RowCount, TRUE),
b = sample.int(9, RowCount, TRUE),
c = sample.int(9, RowCount, TRUE),
d = sample.int(9, RowCount, TRUE),
e = sample.int(9, RowCount, TRUE),
f = sample.int(9, RowCount, TRUE))
## Generate an expression to paste an arbitrary list of columns together
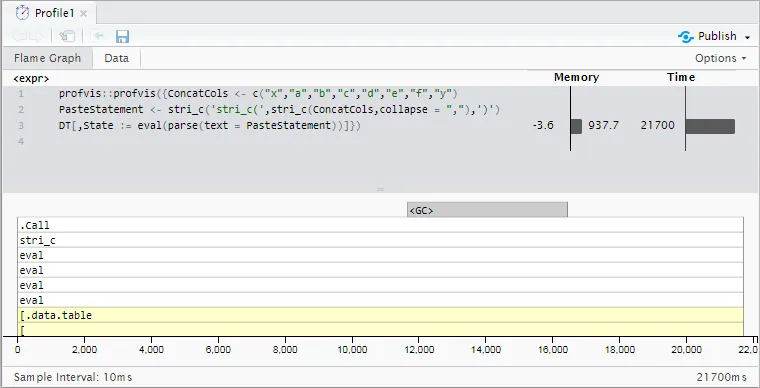
ConcatCols <- c("x","a","b","c","d","e","f","y")
PasteStatement <- stri_c('stri_c(',stri_c(ConcatCols,collapse = ","),')')
print(PasteStatement)
提供
[1] "stri_c(x,a,b,c,d,e,f,y)"
然后使用以下表达式将列连接起来:
DT[,State := eval(parse(text = PasteStatement))]
输出的示例:
x y a b c d e f State
1: foo bar 4 8 3 6 9 2 foo483692bar
2: foo bar 8 4 8 7 8 4 foo848784bar
3: foo bar 2 6 2 4 3 5 foo262435bar
4: foo bar 2 4 2 4 9 9 foo242499bar
5: foo bar 5 9 8 7 2 7 foo598727bar
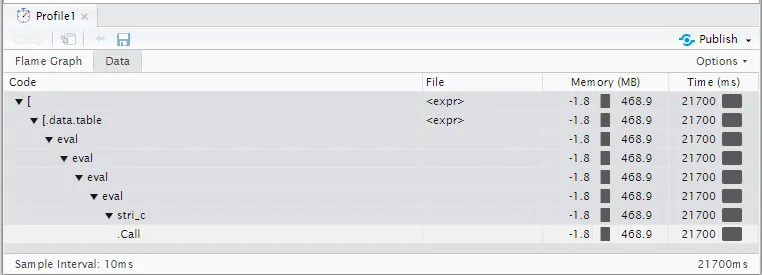
性能分析结果
更新1:fread、fwrite和sed
根据@Gregor的建议,尝试使用sed在磁盘上进行连接。由于data.table的极快速度的fread和fwrite函数,我能够将列写入磁盘,在磁盘上使用sed消除逗号分隔符,然后在约18.3秒内读回后处理过的输出——虽然还不足以使转换,但也是一个有趣的侧面研究!
ConcatCols <- c("x","a","b","c","d","e","f","y")
fwrite(DT[,..ConcatCols],"/home/xxx/DT.csv")
system("sed 's/,//g' /home/xxx/DT.csv > /home/xxx/DT_Post.csv ")
Post <- fread("/home/xxx/DT_Post.csv")
DT[,State := Post[[1]]]
18.3秒的细项分解(无法使用profvis,因为sed对R分析器不可见):
data.table::fwrite()- 0.5秒sed- 14.8秒data.table::fread()- 3.0秒:=- 0.0秒
如果没有其他问题,这证明了data.table作者在磁盘IO性能优化方面的广泛工作。 (我正在使用添加了多线程支持的1.10.5开发版本的fread,fwrite已经有一段时间支持多线程)。
一个注意事项:如果有一种方法可以使用空白分隔符通过fwrite写入文件,就像下面@Gregor建议的那样,那么此方法可能被削减到~3.5秒!
关于这个话题的更新:forked data.table并注释掉需要大于长度0的分隔符的行,神奇地得到了一些空格?在试图搞乱C内部时引起了一些segfaults,所以暂时放一放。理想的解决方案不需要写入磁盘,并且可以将所有内容保存在内存中。
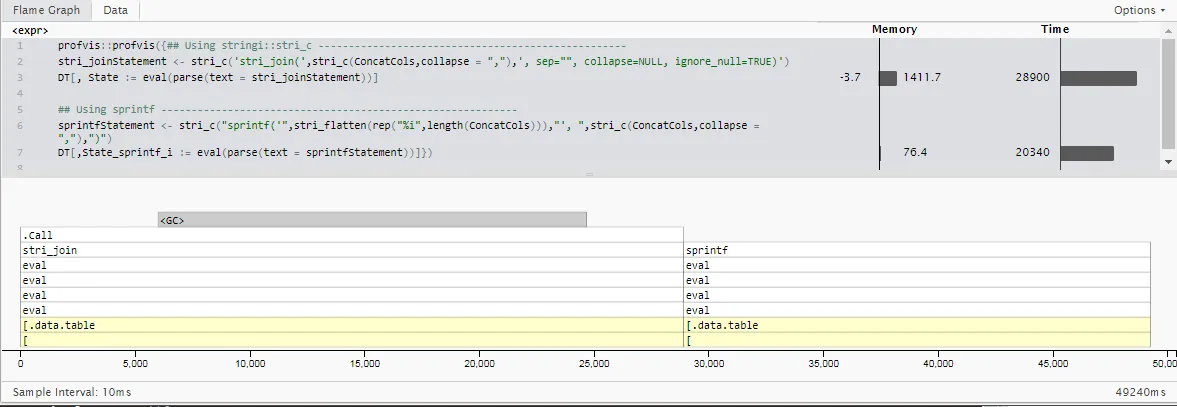
第二次更新:sprintf用于整数特定情况
这里是第二次更新:虽然我在原始用法示例中包括了字符串,但我的实际用例仅串联整数值(可以基于上游清理步骤始终假设非空)。
由于使用情况高度特定并且与先前发布的时间不同,因此我不会直接将时间与先前发布的时间进行比较。 但是,一个结论是,虽然stringi很好地处理了许多字符编码格式,不需要指定混合向量类型,并且在箱外执行了一堆错误处理,但这确实增加了一些时间。(这对大多数情况可能值得)。
通过使用基本的R的sprintf函数并让它事先知道所有输入都将是整数,我们可以削减计算18个整数列的5百万行的运行时间约30%。(20.3秒而不是28.9秒)
library(data.table)
library(stringi)
RowCount <- 5e6
DT <- data.table(x = "foo",
y = "bar",
a = sample.int(9, RowCount, TRUE),
b = sample.int(9, RowCount, TRUE),
c = sample.int(9, RowCount, TRUE),
d = sample.int(9, RowCount, TRUE),
e = sample.int(9, RowCount, TRUE),
f = sample.int(9, RowCount, TRUE))
## Generate an expression to paste an arbitrary list of columns together
ConcatCols <- list("a","b","c","d","e","f")
## Do it 3x as many times
ConcatCols <- c(ConcatCols,ConcatCols,ConcatCols)
## Using stringi::stri_c ---------------------------------------------------
stri_joinStatement <- stri_c('stri_join(',stri_c(ConcatCols,collapse = ","),', sep="", collapse=NULL, ignore_null=TRUE)')
DT[, State := eval(parse(text = stri_joinStatement))]
## Using sprintf -----------------------------------------------------------
sprintfStatement <- stri_c("sprintf('",stri_flatten(rep("%i",length(ConcatCols))),"', ",stri_c(ConcatCols,collapse = ","),")")
DT[,State_sprintf_i := eval(parse(text = sprintfStatement))]
> cat(stri_joinStatement)
stri_join(a,b,c,d,e,f,a,b,c,d,e,f,a,b,c,d,e,f, sep="", collapse=NULL, ignore_null=TRUE)
> cat(sprintfStatement)
sprintf('%i%i%i%i%i%i%i%i%i%i%i%i%i%i%i%i%i%i', a,b,c,d,e,f,a,b,c,d,e,f,a,b,c,d,e,f)
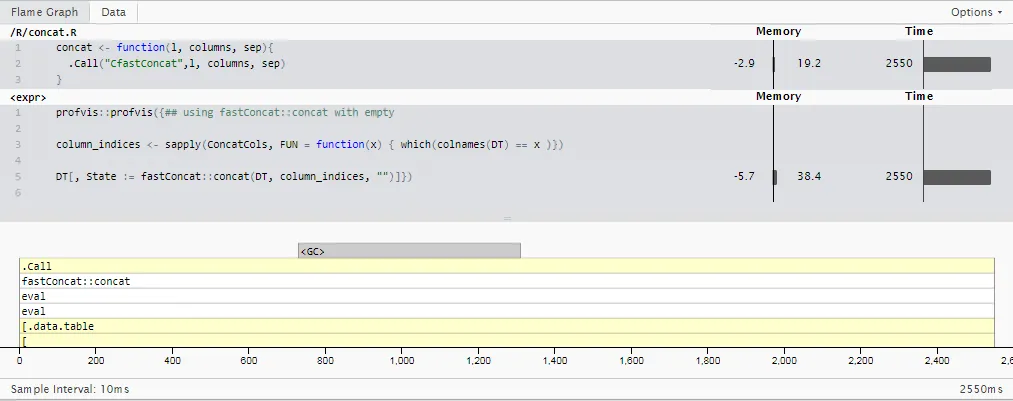
更新 3: R 不必慢。
基于@Martin Modrák的答案,我制作了一个“一招鲜”的软件包,基于一些针对特定“单个数字整数”情况的data.table内部专门化: fastConcat。(不要指望它会很快出现在CRAN上,但是您可以自己承担风险从github repo安装并使用,msummersgill/fastConcat。)
也许有人更好地理解c,这可能进一步改善,但目前为止,它正在运行与更新2中相同的用例,在 2.5秒 内--比sprintf()稍快约 8倍,比我最初使用的stringi::stri_c()方法快 11.5倍。
对我来说,这凸显了在某些最简单的操作中提高性能的巨大机会,例如使用更好调整的c进行基本字符串向量连接。 我想人们像@Matt Dowle这样的人已经看到了这一点--如果只是他有时间重写R的所有内容,而不仅仅是data.frame。
stri_c的作用就是立即调用一个 C++ 函数来连接字符串。我认为你不可能在 R 中超越它的性能。即使paste也很快转换为编译代码,因此其性能几乎一样好。 - Gregor Thomasfread不允许空分隔符,但readr::write_delim可以。它可能太慢了,但值得一试。(c)sed可能是从命令行中最快的方法,但是这个问题的答案表明,如果你复制文件而不是直接在原地编辑文件,你可以通过不同的语法特别是获得一些加速。 - Gregor Thomasfwrite中进行一行输入检查可以防止您将""作为分隔符进行指定。您可以尝试使用fixInNamespace来删除该行,并查看是否允许您使用sep = ""进行fwrite。我以前从未使用过fixInNamespace,但应该是可行的。未解决的问题是sep无法为空字符串的更深层次原因是否存在。 - Gregor Thomassep = ""。 - eddi