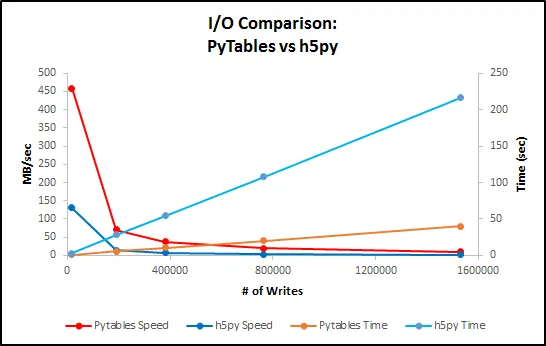
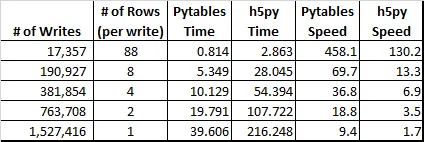
我注意到使用h5py库写入.h5文件比使用pytables库要慢得多。这是为什么?即使数组的形状已知,情况也是如此。此外,我使用相同的块大小和无压缩过滤器。
以下是脚本:
import h5py
import tables
import numpy as np
from time import time
dim1, dim2 = 64, 1527416
# append columns
print("PYTABLES: append columns")
print("=" * 32)
f = tables.open_file("/tmp/test.h5", "w")
a = f.create_earray(f.root, "time_data", tables.Float32Atom(), shape=(0, dim1))
t1 = time()
zeros = np.zeros((1, dim1), dtype="float32")
for i in range(dim2):
a.append(zeros)
tcre = round(time() - t1, 3)
thcre = round(dim1 * dim2 * 4 / (tcre * 1024 * 1024), 1)
print("Time to append %d columns: %s sec (%s MB/s)" % (i+1, tcre, thcre))
print("=" * 32)
chunkshape = a.chunkshape
f.close()
print("H5PY: append columns")
print("=" * 32)
f = h5py.File(name="/tmp/test.h5",mode='w')
a = f.create_dataset(name='time_data',shape=(0, dim1),
maxshape=(None,dim1),dtype='f',chunks=chunkshape)
t1 = time()
zeros = np.zeros((1, dim1), dtype="float32")
samplesWritten = 0
for i in range(dim2):
a.resize((samplesWritten+1, dim1))
a[samplesWritten:(samplesWritten+1),:] = zeros
samplesWritten += 1
tcre = round(time() - t1, 3)
thcre = round(dim1 * dim2 * 4 / (tcre * 1024 * 1024), 1)
print("Time to append %d columns: %s sec (%s MB/s)" % (i+1, tcre, thcre))
print("=" * 32)
f.close()
我的电脑上的返回结果:
PYTABLES: append columns
================================
Time to append 1527416 columns: 22.679 sec (16.4 MB/s)
================================
H5PY: append columns
================================
Time to append 1527416 columns: 158.894 sec (2.3 MB/s)
================================
如果我在每个for循环之后都执行flush操作,例如:
for i in range(dim2):
a.append(zeros)
f.flush()
PYTABLES: append columns
================================
Time to append 1527416 columns: 67.481 sec (5.5 MB/s)
================================
H5PY: append columns
================================
Time to append 1527416 columns: 193.644 sec (1.9 MB/s)
================================