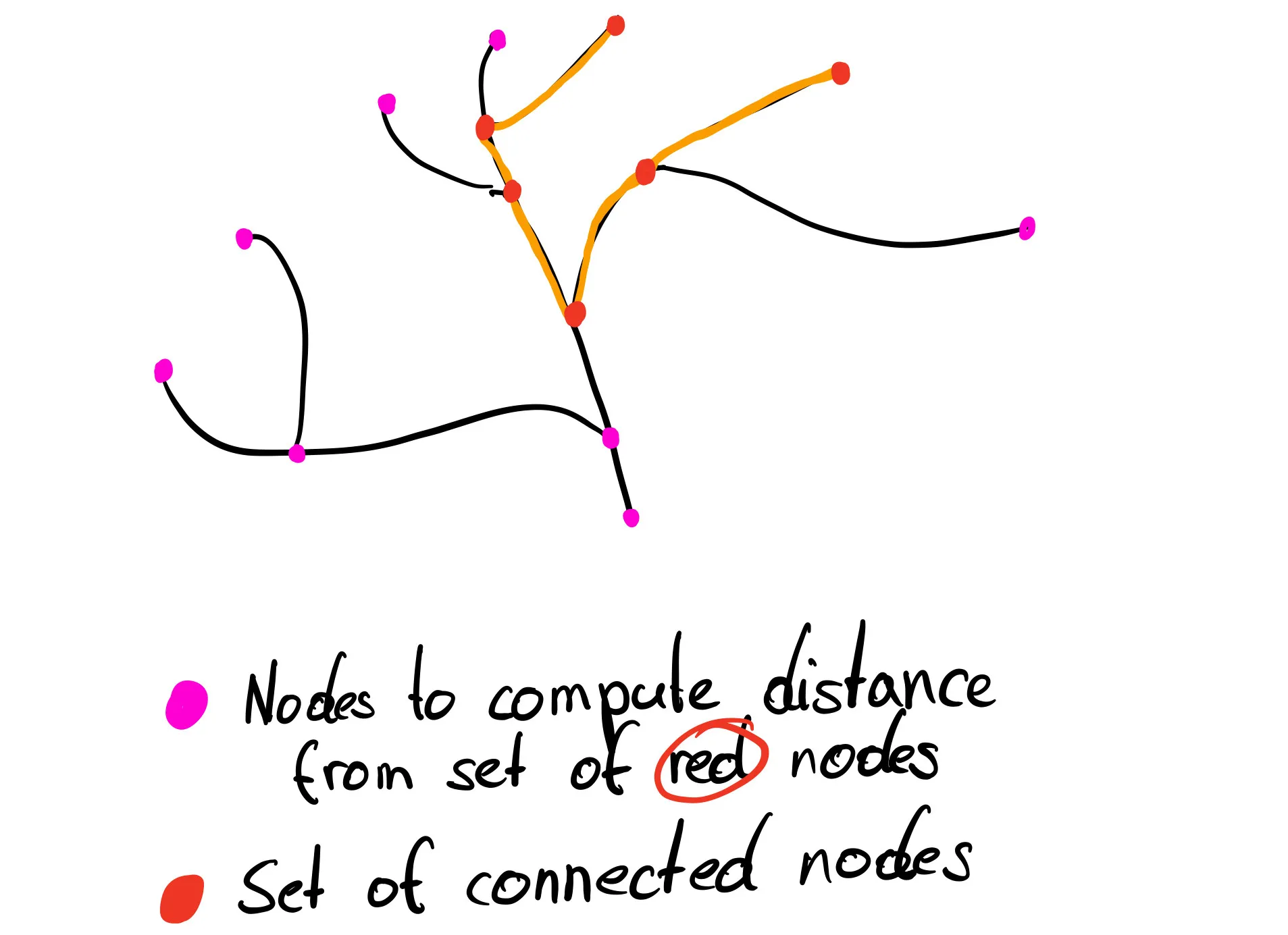
对于无向/无权图中的每个节点(粉色节点),我想计算到给定连接节点集(红色节点)的最近距离。重要的是,计算速度要快,也适用于大型图(~4000个节点)和大量连接节点。请参见下面的示例。哪种算法/图库最适合我做这个?
我已经尝试使用NetworkX - shortest_path_length 来实现类似的功能,但速度太慢了。特别是对于一个大型连接的红节点集合。
import networkx as nx
all_distances = []
for node in red_nodes:
distances = nx.shortest_path_length(graph, source=node) # compute distances to all nodes in the graph from the source node
all_distances.append(distances)
shortest_distances = filter_for_shortest_distance(all_distances)
这是一个关于如何访问我正在使用的图形的示例。红色节点可以是图中任意连通节点的子集。
# Import navis
import navis
# Load one of the example neurons
sk = navis.example_neurons(n=1, kind='skeleton')
# Inspect the neuron graph
graph = sk.get_graph_nx()
navis中的TreeNeurons是有向、无权网络。 - Paul Brodersennx.predecessors,直到遇到红色节点(因为红色节点是混合树的根节点,所以这是有保证的)。 - Paul Brodersen