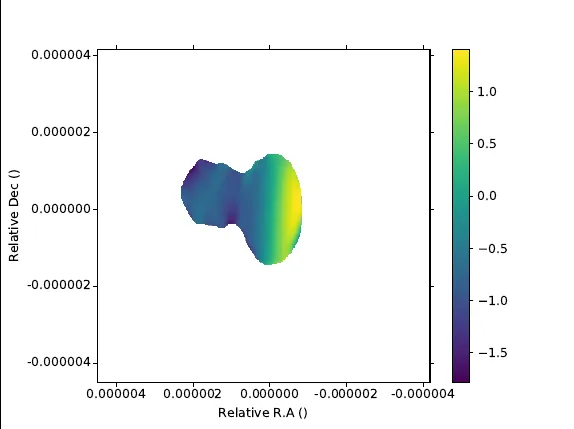
我已经在python中加载并绘制了一个FITS文件。
在之前的帖子的帮助下,我成功地将轴从像素转换为天文坐标。但是我无法正确地将它们转换为毫角秒(mas)。
以下是代码:
输出结果如下图所示: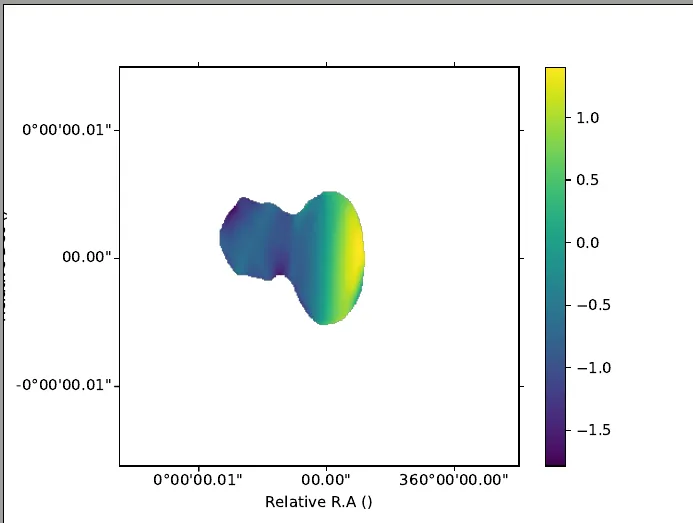 y轴标签因某种原因被切掉了,我不知道为什么。
y轴标签因某种原因被切掉了,我不知道为什么。
正如在另一篇帖子中所示,可以使用
import numpy as np
import matplotlib.pyplot as plt
import astropy.units as u
from astropy.wcs import WCS
from astropy.io import fits
from astropy.utils.data import get_pkg_data_filename
filename = get_pkg_data_filename('hallo.fits')
hdu = fits.open(filename)[0]
wcs = WCS(hdu.header).celestial
wcs.wcs.crval = [0,0]
plt.subplot(projection=wcs)
plt.imshow(hdu.data[0][0], origin='lower')
plt.xlim(200,800)
plt.ylim(200,800)
plt.xlabel('Relative R.A ()')
plt.ylabel('Relative Dec ()')
plt.colorbar()
输出结果如下图所示:
 y轴标签因某种原因被切掉了,我不知道为什么。
y轴标签因某种原因被切掉了,我不知道为什么。正如在另一篇帖子中所示,可以使用
wcs.wcs.ctype = [ 'XOFFSET' , 'YOFFSET' ]
将其转换为毫秒,并得到结果:
但比例尺不正确!例如,0度00分00.02秒应该是20毫角秒,而不是0.000002!我有遗漏什么吗?