我急需一个在Python中使用LibSVM进行分类任务的示例。我不知道输入应该是什么样子,以及哪个函数负责训练和哪个函数负责测试。
这里列出的代码示例不适用于LibSVM 3.1,因此我大致上移植了mossplix的示例:
from svmutil import *
svm_model.predict = lambda self, x: svm_predict([0], [x], self)[0][0]
prob = svm_problem([1,-1], [[1,0,1], [-1,0,-1]])
param = svm_parameter()
param.kernel_type = LINEAR
param.C = 10
m=svm_train(prob, param)
m.predict([1,1,1])
这个示例演示了一个单类支持向量机分类器;它尽可能简单,同时仍然展示了完整的LIBSVM工作流程。
第一步:导入NumPy和LIBSVM。
import numpy as NP
from svm import *
第二步:生成合成数据:对于本示例,生成在给定边界内的500个点(请注意:LIBSVM 网站提供了相当多的真实数据集)。
Data = NP.random.randint(-5, 5, 1000).reshape(500, 2)
步骤 3: 现在,为一个单类分类器选择一些非线性决策边界:
rx = [ (x**2 + y**2) < 9 and 1 or 0 for (x, y) in Data ]
步骤4:接下来,任意划分数据以及这个决策边界有关的内容:
类别I:落在或位于任意圆形内部的那些数据点
类别II:所有决策边界(圆形)外面的点
SVM模型构建从这里开始;在此之前的所有步骤只是为了准备一些合成数据。
步骤5:通过调用svm_problem构建问题描述,将决策边界函数和数据传递给它,然后将结果绑定到一个变量上。
px = svm_problem(rx, Data)
第六步:选择非线性映射的核函数
对于这个例子,我选择了RBF(径向基函数)作为我的核函数。
pm = svm_parameter(kernel_type=RBF)
第7步:通过调用svm_model,传递问题描述(px)和核函数(pm)来训练分类器。
v = svm_model(px, pm)
第八步:最后,通过在训练好的模型对象('v')上调用predict函数来测试训练好的分类器。
v.predict([3, 1])
# returns the class label (either '1' or '0')
关于您问题中有关选择核函数的部分,支持向量机并不特定于某个核函数 - 例如,我可以选择不同的核函数(高斯、多项式等)。
LIBSVM 包括所有最常用的核函数-这是一个很大的帮助,因为您可以看到所有可能的替代方案,并选择一个用于模型的核函数只是调用 svm_parameter 并传递一个值给 kernel_type(所选核函数的三字母缩写)。
最后,您选择用于训练的核函数必须与对测试数据使用的核函数匹配。
3
LIBSVM从包含两个列表的元组中读取数据。第一个列表包含类别,第二个列表包含输入数据。使用两个可能的类创建简单数据集,您还需要通过创建svm_parameter指定要使用的核函数。
>> from libsvm import *
# 导入libsvm库
>> prob = svm_problem([1,-1],[[1,0,1],[-1,0,-1]])
# 创建SVM问题实例,指定标签(1,-1)和训练数据(特征向量)
>> param = svm_parameter(kernel_type = LINEAR, C = 10)
# 创建SVM参数实例,指定使用线性核函数和惩罚参数C为10
# 训练模型
>> m = svm_model(prob, param)
# 测试模型
>> m.predict([1, 1, 1])
# 预测新数据的分类结果
2
补充@shinNoNoir的内容:
param.kernel_type表示你想使用的内核函数类型, 0:线性 1:多项式 2:径向基函数 3:Sigmoid
另外请注意,svm_problem(y, x):这里y是类标签,x是类实例,x和y只能是列表、元组和字典(不能是numpy数组)。
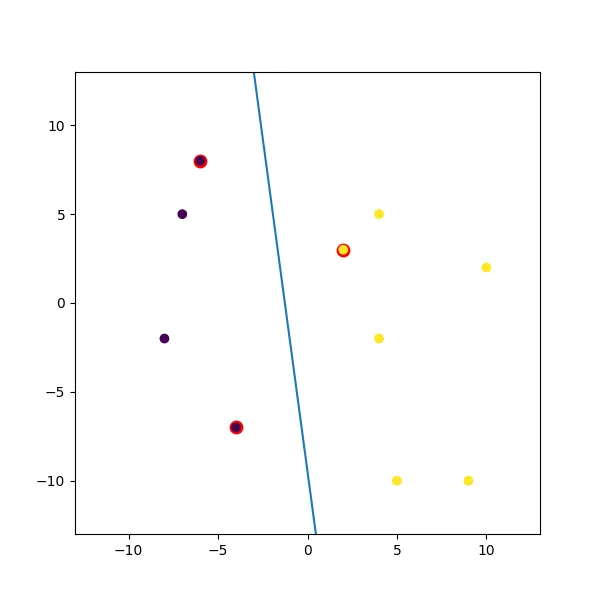
这里是一个我混合在一起的虚拟示例:
import numpy
import matplotlib.pyplot as plt
from random import seed
from random import randrange
import svmutil as svm
seed(1)
# Creating Data (Dense)
train = list([randrange(-10, 11), randrange(-10, 11)] for i in range(10))
labels = [-1, -1, -1, 1, 1, -1, 1, 1, 1, 1]
options = '-t 0' # linear model
# Training Model
model = svm.svm_train(labels, train, options)
# Line Parameters
w = numpy.matmul(numpy.array(train)[numpy.array(model.get_sv_indices()) - 1].T, model.get_sv_coef())
b = -model.rho.contents.value
if model.get_labels()[1] == -1: # No idea here but it should be done :|
w = -w
b = -b
print(w)
print(b)
# Plotting
plt.figure(figsize=(6, 6))
for i in model.get_sv_indices():
plt.scatter(train[i - 1][0], train[i - 1][1], color='red', s=80)
train = numpy.array(train).T
plt.scatter(train[0], train[1], c=labels)
plt.plot([-5, 5], [-(-5 * w[0] + b) / w[1], -(5 * w[0] + b) / w[1]])
plt.xlim([-13, 13])
plt.ylim([-13, 13])
plt.show()
使用Scikit-learn进行SVM:
from sklearn.svm import SVC
X = [[0, 0], [1, 1]]
y = [0, 1]
model = SVC().fit(X, y)
tests = [[0.,0.], [0.49,0.49], [0.5,0.5], [2., 2.]]
print(model.predict(tests))
# prints [0 0 1 1]
param = svm_parameter('-s 0 -t 2 -d 3 -c '+str(C)+' -g '+str(G)+' -p '+str(self.epsilon)+' -n '+str(self.nu))
我不知道早期版本的情况,但在LibSVM 3.xx中,方法
svm_parameter('options')只需要一个参数。在我的情况下,
C、G、p和nu是动态值。您可以根据您的代码进行更改。
选项:
-s svm_type : set type of SVM (default 0)
0 -- C-SVC (multi-class classification)
1 -- nu-SVC (multi-class classification)
2 -- one-class SVM
3 -- epsilon-SVR (regression)
4 -- nu-SVR (regression)
-t kernel_type : set type of kernel function (default 2)
0 -- linear: u'*v
1 -- polynomial: (gamma*u'*v + coef0)^degree
2 -- radial basis function: exp(-gamma*|u-v|^2)
3 -- sigmoid: tanh(gamma*u'*v + coef0)
4 -- precomputed kernel (kernel values in training_set_file)
-d degree : set degree in kernel function (default 3)
-g gamma : set gamma in kernel function (default 1/num_features)
-r coef0 : set coef0 in kernel function (default 0)
-c cost : set the parameter C of C-SVC, epsilon-SVR, and nu-SVR (default 1)
-n nu : set the parameter nu of nu-SVC, one-class SVM, and nu-SVR (default 0.5)
-p epsilon : set the epsilon in loss function of epsilon-SVR (default 0.1)
-m cachesize : set cache memory size in MB (default 100)
-e epsilon : set tolerance of termination criterion (default 0.001)
-h shrinking : whether to use the shrinking heuristics, 0 or 1 (default 1)
-b probability_estimates : whether to train a SVC or SVR model for probability estimates, 0 or 1 (default 0)
-wi weight : set the parameter C of class i to weight*C, for C-SVC (default 1)
-v n: n-fold cross validation mode
-q : quiet mode (no outputs)
原文链接
Traceback (most recent call last): File "<stdin>", line 1, in <module> File "/usr/lib/pymodules/python2.7/svm.py", line 83, in __init__ tmp_xi, tmp_idx = gen_svm_nodearray(xi,isKernel=isKernel) File "/usr/lib/pymodules/python2.7/svm.py", line 51, in gen_svm_nodearray raise TypeError('xi should be a dictionary, list or tuple') TypeError: xi should be a dictionary, list or tuple。该错误意味着在svm.py文件中的第83行,在初始化函数时无法将输入(xi)转换为正确的形式。xi应该是字典、列表或元组类型。 - cnvzmxcvmcx