对于具有
0..*:0..1 基数的左连接或具有
0..1:0..* 基数的右连接,可以直接将连接器(
0..1 表)的单边列分配到被连接者(
0..* 表)中,从而避免创建一个全新的数据表。这需要将被连接者的关键列与连接器进行匹配,并根据赋值索引 + 排序连接器的行。
如果关键字是单个列,则我们可以使用单个调用
match() 进行匹配。这是我在本答案中要介绍的情况。
以下是基于 OP 的示例,除此之外,我还添加了一个 id 为 7 的额外行到
df2 中,以测试连接器中不存在匹配键的情况。这实际上是
df1 左连接
df2:
df1 <- data.frame(CustomerId=1:6,Product=c(rep('Toaster',3L),rep('Radio',3L)));
df2 <- data.frame(CustomerId=c(2L,4L,6L,7L),State=c(rep('Alabama',2L),'Ohio','Texas'));
df1[names(df2)[-1L]] <- df2[match(df1[,1L],df2[,1L]),-1L];
df1;
在上面的代码中,我硬编码了一个假设,即关键列是两个输入表格的第一列。我认为,一般情况下,这并不是一个不合理的假设,因为如果你有一个带有关键列的数据框,如果它没有从一开始就被设置为数据框的第一列,那么这将是奇怪的。而且你可以重新排序列来使其成为第一列。这种假设的一个优点是关键列的名称不必是硬编码的,虽然我想这只是用另一个假设替换了一个假设。整数索引的简洁性和速度是另一个优点,在下面的基准测试中,我将更改实现以使用字符串名称索引,以匹配竞争实现。
我认为,如果你有几个表格想要与单个大表进行左连接,那么这是一个特别合适的解决方案。对于每个合并重建整个表格是不必要和低效的。
另一方面,如果你需要 joinee 保持不变,无论出于什么原因,在这个操作中,这个解决方案不能被使用,因为它直接修改了 joinee。尽管在这种情况下,你可以简单地复制并对副本执行原地赋值操作。
作为一个旁注,我简要研究了多列键的可能匹配解决方案。不幸的是,我发现的唯一匹配解决方案是:
- 低效的连接,例如
match(interaction(df1$a,df1$b),interaction(df2$a,df2$b)),或者使用
paste()相同的想法。
- 低效的笛卡尔连接,例如
outer(df1$a,df2$a,`==`) & outer(df1$b,df2$b,`==`)。
- 基于基础R
merge()和等效的基于包的合并函数,它们总是分配一个新表来返回合并的结果,因此不适合于基于就地赋值的解决方案。
例如,查看
在不同数据框上匹配多列并获取其他列作为结果,
将两列与另外两列进行匹配,
在多个列上匹配, 以及我最初提出就地解决方案的这个问题的重复项,
在R中合并具有不同行数的两个数据框。
基准测试
我决定自己进行基准测试,以比较原地赋值方法与其他已提供的解决方案在此问题中的差异。
测试代码:
library(microbenchmark);
library(data.table);
library(sqldf);
library(plyr);
library(dplyr);
solSpecs <- list(
merge=list(testFuncs=list(
inner=function(df1,df2,key) merge(df1,df2,key),
left =function(df1,df2,key) merge(df1,df2,key,all.x=T),
right=function(df1,df2,key) merge(df1,df2,key,all.y=T),
full =function(df1,df2,key) merge(df1,df2,key,all=T)
)),
data.table.unkeyed=list(argSpec='data.table.unkeyed',testFuncs=list(
inner=function(dt1,dt2,key) dt1[dt2,on=key,nomatch=0L,allow.cartesian=T],
left =function(dt1,dt2,key) dt2[dt1,on=key,allow.cartesian=T],
right=function(dt1,dt2,key) dt1[dt2,on=key,allow.cartesian=T],
full =function(dt1,dt2,key) merge(dt1,dt2,key,all=T,allow.cartesian=T)
)),
data.table.keyed=list(argSpec='data.table.keyed',testFuncs=list(
inner=function(dt1,dt2) dt1[dt2,nomatch=0L,allow.cartesian=T],
left =function(dt1,dt2) dt2[dt1,allow.cartesian=T],
right=function(dt1,dt2) dt1[dt2,allow.cartesian=T],
full =function(dt1,dt2) merge(dt1,dt2,all=T,allow.cartesian=T)
)),
sqldf.unindexed=list(testFuncs=list(
inner=function(df1,df2,key) sqldf(paste0('select * from df1 inner join df2 using(',paste(collapse=',',key),')'),connection=NULL),
left =function(df1,df2,key) sqldf(paste0('select * from df1 left join df2 using(',paste(collapse=',',key),')'),connection=NULL),
right=function(df1,df2,key) sqldf(paste0('select * from df2 left join df1 using(',paste(collapse=',',key),')'),connection=NULL)
)),
sqldf.indexed=list(testFuncs=list(
inner=function(df1,df2,key) sqldf(paste0('select * from main.df1 inner join main.df2 using(',paste(collapse=',',key),')')),
left =function(df1,df2,key) sqldf(paste0('select * from main.df1 left join main.df2 using(',paste(collapse=',',key),')')),
right=function(df1,df2,key) sqldf(paste0('select * from main.df2 left join main.df1 using(',paste(collapse=',',key),')'))
)),
plyr=list(testFuncs=list(
inner=function(df1,df2,key) join(df1,df2,key,'inner'),
left =function(df1,df2,key) join(df1,df2,key,'left'),
right=function(df1,df2,key) join(df1,df2,key,'right'),
full =function(df1,df2,key) join(df1,df2,key,'full')
)),
dplyr=list(testFuncs=list(
inner=function(df1,df2,key) inner_join(df1,df2,key),
left =function(df1,df2,key) left_join(df1,df2,key),
right=function(df1,df2,key) right_join(df1,df2,key),
full =function(df1,df2,key) full_join(df1,df2,key)
)),
in.place=list(testFuncs=list(
left =function(df1,df2,key) { cns <- setdiff(names(df2),key); df1[cns] <- df2[match(df1[,key],df2[,key]),cns]; df1; },
right=function(df1,df2,key) { cns <- setdiff(names(df1),key); df2[cns] <- df1[match(df2[,key],df1[,key]),cns]; df2; }
))
);
getSolTypes <- function() names(solSpecs);
getJoinTypes <- function() unique(unlist(lapply(solSpecs,function(x) names(x$testFuncs))));
getArgSpec <- function(argSpecs,key=NULL) if (is.null(key)) argSpecs$default else argSpecs[[key]];
initSqldf <- function() {
sqldf();
if (exists('sqldfInitFlag',envir=globalenv(),inherits=F) && sqldfInitFlag) {
sqldf();
} else {
assign('sqldfInitFlag',T,envir=globalenv());
};
invisible();
};
setUpBenchmarkCall <- function(argSpecs,joinType,solTypes=getSolTypes(),env=parent.frame()) {
callExpressions <- list();
nms <- character();
for (solType in solTypes) {
testFunc <- solSpecs[[solType]]$testFuncs[[joinType]];
if (is.null(testFunc)) next;
testFuncName <- paste0('tf.',solType);
assign(testFuncName,testFunc,envir=env);
argSpecKey <- solSpecs[[solType]]$argSpec;
argSpec <- getArgSpec(argSpecs,argSpecKey);
argList <- setNames(nm=names(argSpec$args),vector('list',length(argSpec$args)));
for (i in seq_along(argSpec$args)) {
argName <- paste0('tfa.',argSpecKey,i);
assign(argName,argSpec$args[[i]],envir=env);
argList[[i]] <- if (i%in%argSpec$copySpec) call('copy',as.symbol(argName)) else as.symbol(argName);
};
callExpressions[[length(callExpressions)+1L]] <- do.call(call,c(list(testFuncName),argList),quote=T);
nms[length(nms)+1L] <- solType;
};
names(callExpressions) <- nms;
callExpressions;
};
harmonize <- function(res) {
res <- as.data.frame(res);
for (ci in which(sapply(res,is.factor))) res[[ci]] <- as.character(res[[ci]]);
for (ci in which(sapply(res,is.logical))) res[[ci]] <- as.integer(res[[ci]]);
res <- res[order(names(res))];
res <- res[do.call(order,res),];
res;
};
checkIdentical <- function(argSpecs,solTypes=getSolTypes()) {
for (joinType in getJoinTypes()) {
callExpressions <- setUpBenchmarkCall(argSpecs,joinType,solTypes);
if (length(callExpressions)<2L) next;
ex <- harmonize(eval(callExpressions[[1L]]));
for (i in seq(2L,len=length(callExpressions)-1L)) {
y <- harmonize(eval(callExpressions[[i]]));
if (!isTRUE(all.equal(ex,y,check.attributes=F))) {
ex <<- ex;
y <<- y;
solType <- names(callExpressions)[i];
stop(paste0('non-identical: ',solType,' ',joinType,'.'));
};
};
};
invisible();
};
testJoinType <- function(argSpecs,joinType,solTypes=getSolTypes(),metric=NULL,times=100L) {
callExpressions <- setUpBenchmarkCall(argSpecs,joinType,solTypes);
bm <- microbenchmark(list=callExpressions,times=times);
if (is.null(metric)) return(bm);
bm <- summary(bm);
res <- setNames(nm=names(callExpressions),bm[[metric]]);
attr(res,'unit') <- attr(bm,'unit');
res;
};
testAllJoinTypes <- function(argSpecs,solTypes=getSolTypes(),metric=NULL,times=100L) {
joinTypes <- getJoinTypes();
resList <- setNames(nm=joinTypes,lapply(joinTypes,function(joinType) testJoinType(argSpecs,joinType,solTypes,metric,times)));
if (is.null(metric)) return(resList);
units <- unname(unlist(lapply(resList,attr,'unit')));
res <- do.call(data.frame,c(list(join=joinTypes),setNames(nm=solTypes,rep(list(rep(NA_real_,length(joinTypes))),length(solTypes))),list(unit=units,stringsAsFactors=F)));
for (i in seq_along(resList)) res[i,match(names(resList[[i]]),names(res))] <- resList[[i]];
res;
};
testGrid <- function(makeArgSpecsFunc,sizes,overlaps,solTypes=getSolTypes(),joinTypes=getJoinTypes(),metric='median',times=100L) {
res <- expand.grid(size=sizes,overlap=overlaps,joinType=joinTypes,stringsAsFactors=F);
res[solTypes] <- NA_real_;
res$unit <- NA_character_;
for (ri in seq_len(nrow(res))) {
size <- res$size[ri];
overlap <- res$overlap[ri];
joinType <- res$joinType[ri];
argSpecs <- makeArgSpecsFunc(size,overlap);
checkIdentical(argSpecs,solTypes);
cur <- testJoinType(argSpecs,joinType,solTypes,metric,times);
res[ri,match(names(cur),names(res))] <- cur;
res$unit[ri] <- attr(cur,'unit');
};
res;
};
这是一个基于之前我演示的 OP 示例的基准测试结果:
argSpecs <- list(
default=list(copySpec=1:2,args=list(
df1 <- data.frame(CustomerId=1:6,Product=c(rep('Toaster',3L),rep('Radio',3L))),
df2 <- data.frame(CustomerId=c(2L,4L,6L,7L),State=c(rep('Alabama',2L),'Ohio','Texas')),
'CustomerId'
)),
data.table.unkeyed=list(copySpec=1:2,args=list(
as.data.table(df1),
as.data.table(df2),
'CustomerId'
)),
data.table.keyed=list(copySpec=1:2,args=list(
setkey(as.data.table(df1),CustomerId),
setkey(as.data.table(df2),CustomerId)
))
);
initSqldf();
sqldf('create index df1_key on df1(CustomerId);');
sqldf('create index df2_key on df2(CustomerId);');
checkIdentical(argSpecs);
testAllJoinTypes(argSpecs,metric='median');
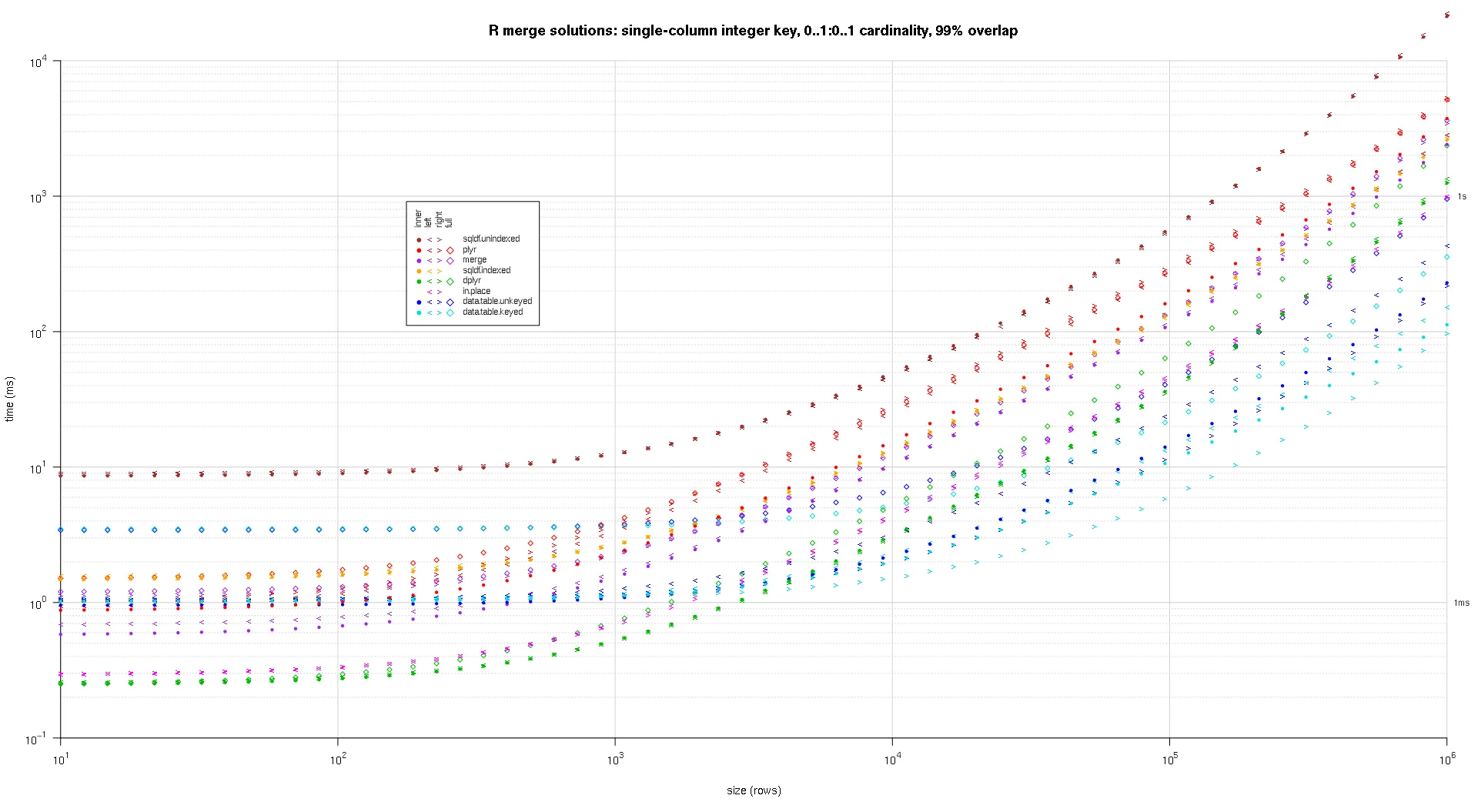
在这里,我对随机输入数据进行基准测试,尝试不同的比例和两个输入表之间关键重叠的不同模式。这个基准测试仍然局限于单列整数键的情况。此外,为了确保原地解决方案适用于相同表格的左连接和右连接,所有随机测试数据使用0..1:0..1基数。这是通过在生成第二个数据框的关键列时无替换抽样第一个数据框的关键列来实现的。
makeArgSpecs.singleIntegerKey.optionalOneToOne <- function(size,overlap) {
com <- as.integer(size*overlap);
argSpecs <- list(
default=list(copySpec=1:2,args=list(
df1 <- data.frame(id=sample(size),y1=rnorm(size),y2=rnorm(size)),
df2 <- data.frame(id=sample(c(if (com>0L) sample(df1$id,com) else integer(),seq(size+1L,len=size-com))),y3=rnorm(size),y4=rnorm(size)),
'id'
)),
data.table.unkeyed=list(copySpec=1:2,args=list(
as.data.table(df1),
as.data.table(df2),
'id'
)),
data.table.keyed=list(copySpec=1:2,args=list(
setkey(as.data.table(df1),id),
setkey(as.data.table(df2),id)
))
);
initSqldf();
sqldf('create index df1_key on df1(id);');
sqldf('create index df2_key on df2(id);');
argSpecs;
};
sizes <- c(1e1L,1e3L,1e6L);
overlaps <- c(0.99,0.5,0.01);
system.time({ res <- testGrid(makeArgSpecs.singleIntegerKey.optionalOneToOne,sizes,overlaps); });
我编写了一些代码来创建上述结果的对数对数图。我为每个重叠百分比生成了一个单独的图。虽然有点混乱,但我喜欢在同一个图中表示所有的解决方案类型和连接类型。
我使用样条插值来显示每种解决方案/连接类型组合的平滑曲线,并用独立的pch符号绘制。连接类型由pch符号捕获,使用点表示内部,左右角括号表示左右,而菱形表示完整。解决方案类型由颜色表示,如图例所示。
plotRes <- function(res,titleFunc,useFloor=F) {
solTypes <- setdiff(names(res),c('size','overlap','joinType','unit'));
normMult <- c(microseconds=1e-3,milliseconds=1);
joinTypes <- getJoinTypes();
cols <- c(merge='purple',data.table.unkeyed='blue',data.table.keyed='#00DDDD',sqldf.unindexed='brown',sqldf.indexed='orange',plyr='red',dplyr='#00BB00',in.place='magenta');
pchs <- list(inner=20L,left='<',right='>',full=23L);
cexs <- c(inner=0.7,left=1,right=1,full=0.7);
NP <- 60L;
ord <- order(decreasing=T,colMeans(res[res$size==max(res$size),solTypes],na.rm=T));
ymajors <- data.frame(y=c(1,1e3),label=c('1ms','1s'),stringsAsFactors=F);
for (overlap in unique(res$overlap)) {
x1 <- res[res$overlap==overlap,];
x1[solTypes] <- x1[solTypes]*normMult[x1$unit]; x1$unit <- NULL;
xlim <- c(1e1,max(x1$size));
xticks <- 10^seq(log10(xlim[1L]),log10(xlim[2L]));
ylim <- c(1e-1,10^((if (useFloor) floor else ceiling)(log10(max(x1[solTypes],na.rm=T)))));
yticks <- 10^seq(log10(ylim[1L]),log10(ylim[2L]));
yticks.minor <- rep(yticks[-length(yticks)],each=9L)*1:9;
plot(NA,xlim=xlim,ylim=ylim,xaxs='i',yaxs='i',axes=F,xlab='size (rows)',ylab='time (ms)',log='xy');
abline(v=xticks,col='lightgrey');
abline(h=yticks.minor,col='lightgrey',lty=3L);
abline(h=yticks,col='lightgrey');
axis(1L,xticks,parse(text=sprintf('10^%d',as.integer(log10(xticks)))));
axis(2L,yticks,parse(text=sprintf('10^%d',as.integer(log10(yticks)))),las=1L);
axis(4L,ymajors$y,ymajors$label,las=1L,tick=F,cex.axis=0.7,hadj=0.5);
for (joinType in rev(joinTypes)) {
x2 <- x1[x1$joinType==joinType,];
for (solType in solTypes) {
if (any(!is.na(x2[[solType]]))) {
xy <- spline(x2$size,x2[[solType]],xout=10^(seq(log10(x2$size[1L]),log10(x2$size[nrow(x2)]),len=NP)));
points(xy$x,xy$y,pch=pchs[[joinType]],col=cols[solType],cex=cexs[joinType],xpd=NA);
};
};
};
leg.cex <- 0.7;
leg.x.in <- grconvertX(0.275,'nfc','in');
leg.y.in <- grconvertY(0.6,'nfc','in');
leg.x.user <- grconvertX(leg.x.in,'in');
leg.y.user <- grconvertY(leg.y.in,'in');
leg.outpad.w.in <- 0.1;
leg.outpad.h.in <- 0.1;
leg.midpad.w.in <- 0.1;
leg.midpad.h.in <- 0.1;
leg.sol.w.in <- max(strwidth(solTypes,'in',leg.cex));
leg.sol.h.in <- max(strheight(solTypes,'in',leg.cex))*1.5;
leg.join.w.in <- max(strheight(joinTypes,'in',leg.cex))*1.5;
leg.join.h.in <- max(strwidth(joinTypes,'in',leg.cex));
leg.main.w.in <- leg.join.w.in*length(joinTypes);
leg.main.h.in <- leg.sol.h.in*length(solTypes);
leg.x2.user <- grconvertX(leg.x.in+leg.outpad.w.in*2+leg.main.w.in+leg.midpad.w.in+leg.sol.w.in,'in');
leg.y2.user <- grconvertY(leg.y.in+leg.outpad.h.in*2+leg.main.h.in+leg.midpad.h.in+leg.join.h.in,'in');
leg.cols.x.user <- grconvertX(leg.x.in+leg.outpad.w.in+leg.join.w.in*(0.5+seq(0L,length(joinTypes)-1L)),'in');
leg.lines.y.user <- grconvertY(leg.y.in+leg.outpad.h.in+leg.main.h.in-leg.sol.h.in*(0.5+seq(0L,length(solTypes)-1L)),'in');
leg.sol.x.user <- grconvertX(leg.x.in+leg.outpad.w.in+leg.main.w.in+leg.midpad.w.in,'in');
leg.join.y.user <- grconvertY(leg.y.in+leg.outpad.h.in+leg.main.h.in+leg.midpad.h.in,'in');
rect(leg.x.user,leg.y.user,leg.x2.user,leg.y2.user,col='white');
text(leg.sol.x.user,leg.lines.y.user,solTypes[ord],cex=leg.cex,pos=4L,offset=0);
text(leg.cols.x.user,leg.join.y.user,joinTypes,cex=leg.cex,pos=4L,offset=0,srt=90);
for (i in seq_along(joinTypes)) {
joinType <- joinTypes[i];
points(rep(leg.cols.x.user[i],length(solTypes)),ifelse(colSums(!is.na(x1[x1$joinType==joinType,solTypes[ord]]))==0L,NA,leg.lines.y.user),pch=pchs[[joinType]],col=cols[solTypes[ord]]);
};
title(titleFunc(overlap));
readline(sprintf('overlap %.02f',overlap));
};
};
titleFunc <- function(overlap) sprintf('R merge solutions: single-column integer key, 0..1:0..1 cardinality, %d%% overlap',as.integer(overlap*100));
plotRes(res,titleFunc,T);
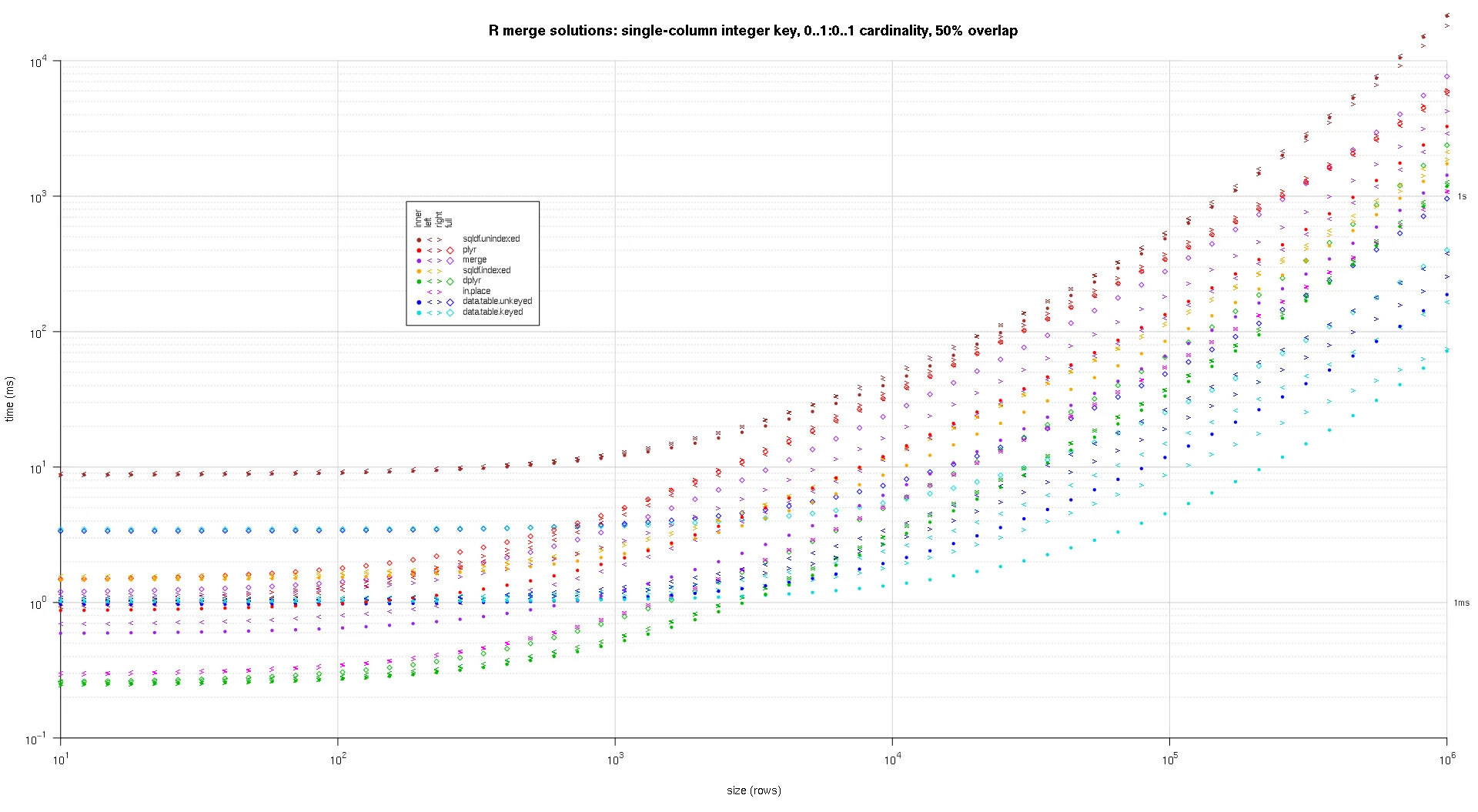
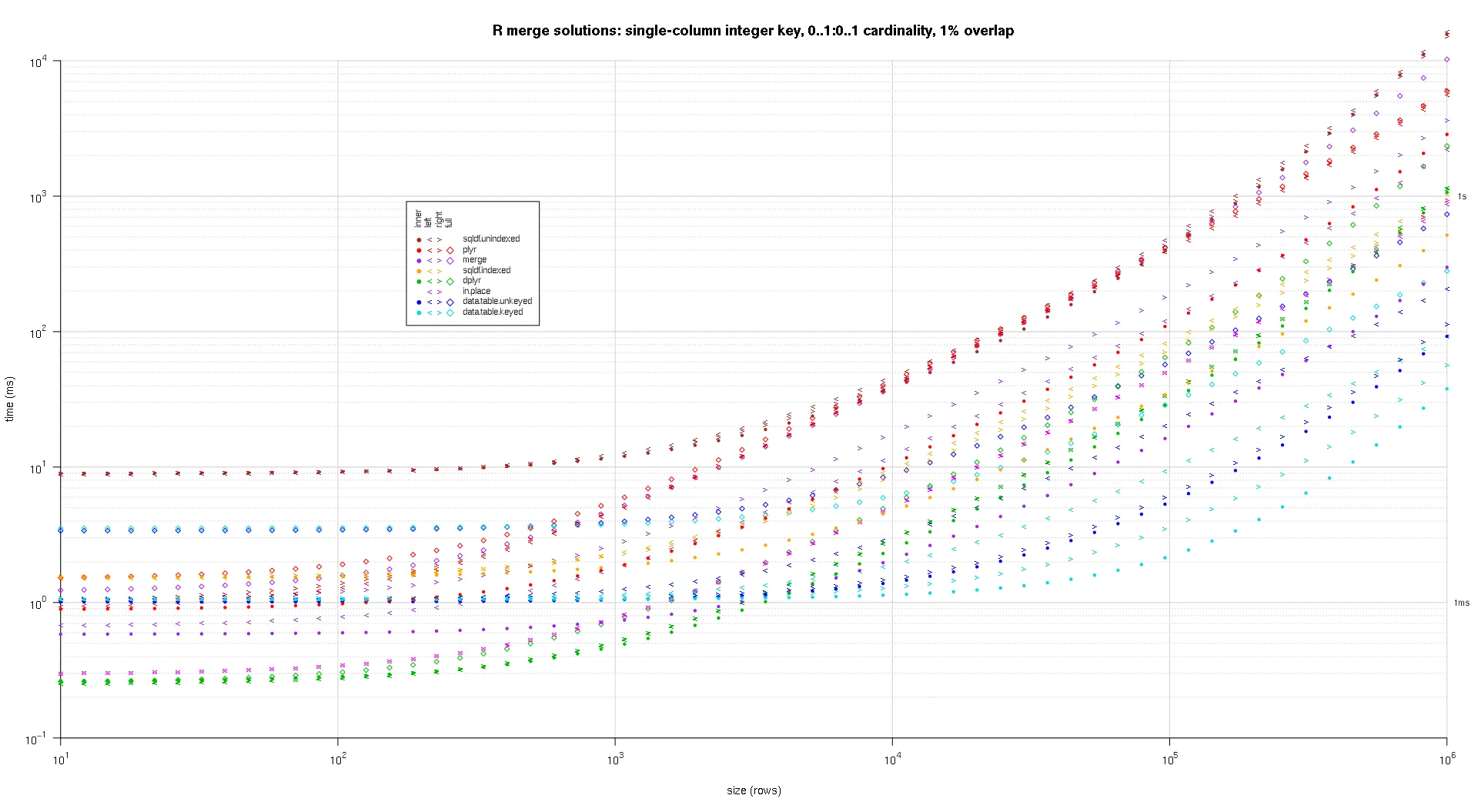
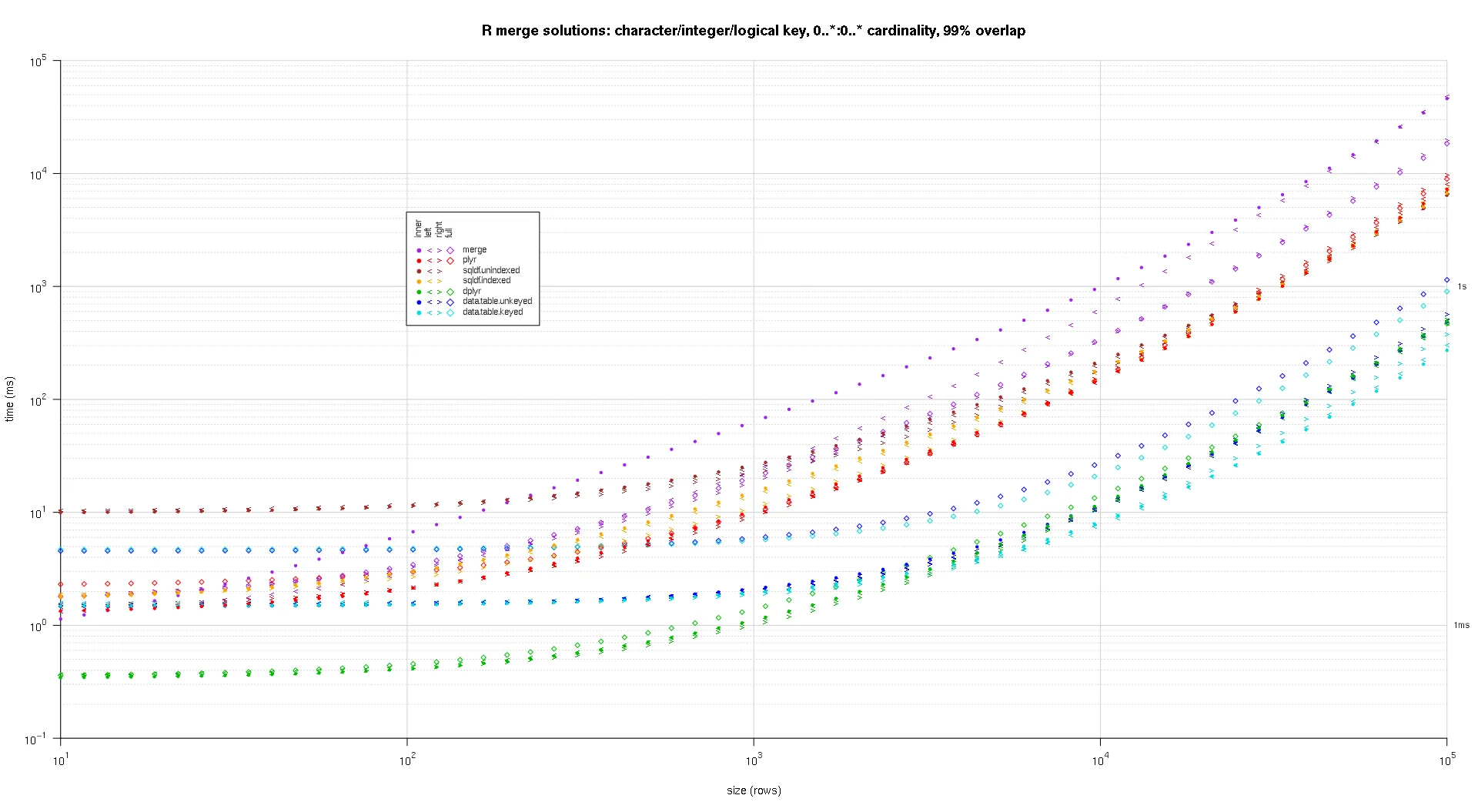
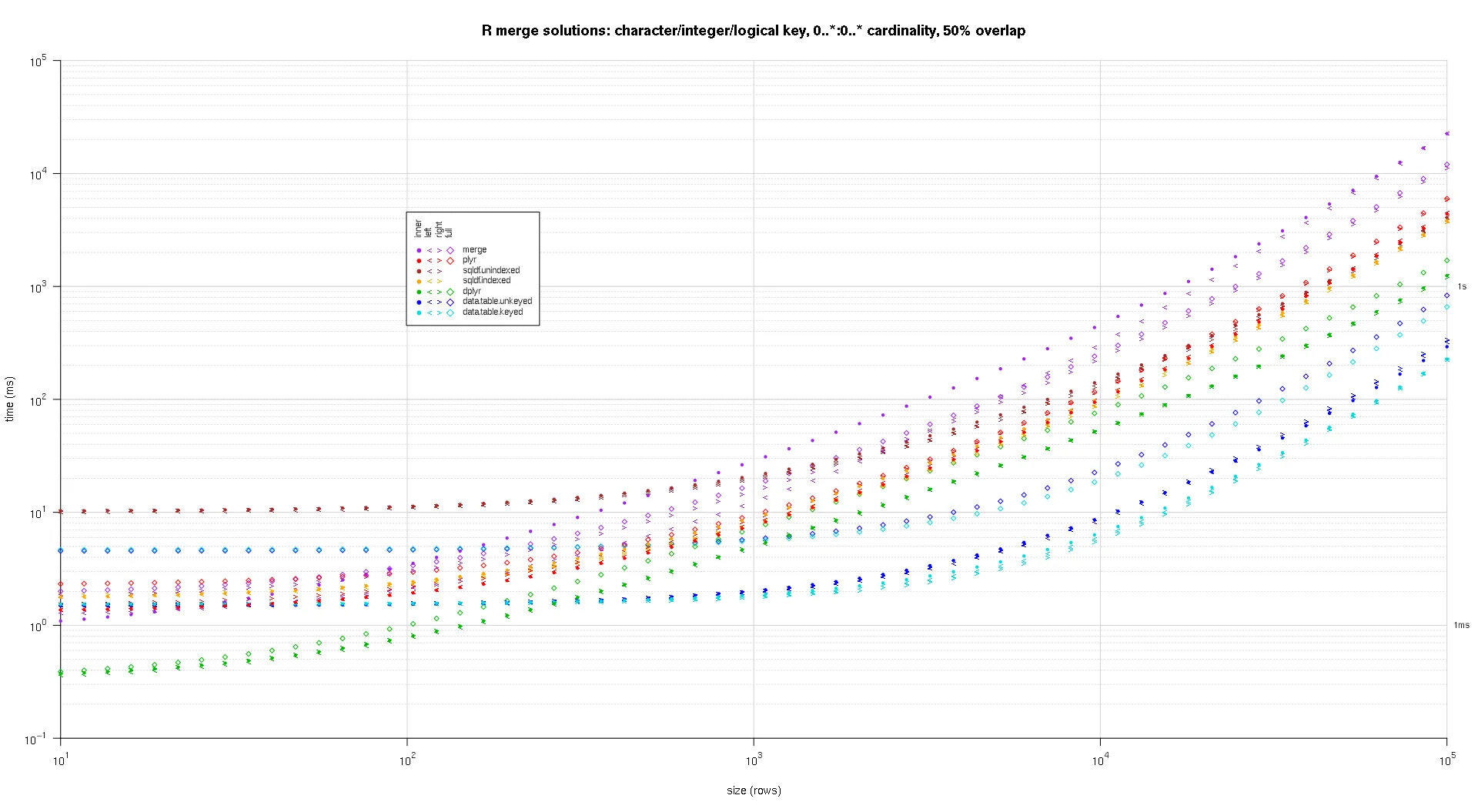
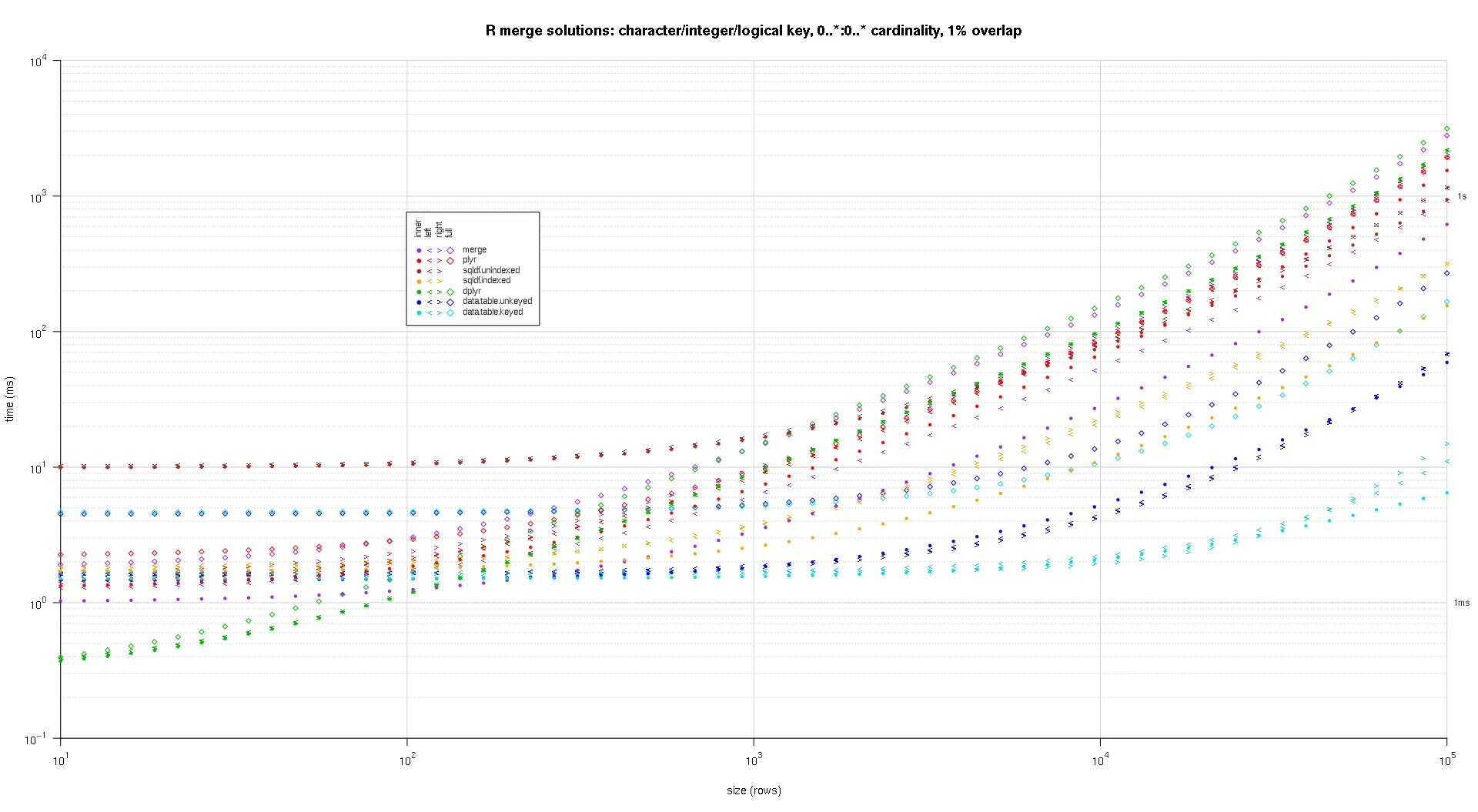
这是第二个大规模基准测试,涉及关键列的数量和类型以及基数等方面更加重型。对于此基准测试,我使用了三个关键列:一个字符、一个整数和一个逻辑值,对基数没有限制(即
0..*:0..*)。(一般来说,不建议定义具有双精度或复杂值的关键列,因为会出现浮点比较问题,而且基本上没有人使用原始类型,更不用说用于关键列了,因此我没有将这些类型包括在关键列中。此外,出于信息考虑,我最初尝试使用四个关键列,其中包括一个 POSIXct 关键列,但由于某种原因,POSIXct 类型与
sqldf.indexed 解决方案不兼容,可能是由于浮点比较异常,因此我将其删除。)
makeArgSpecs.assortedKey.optionalManyToMany <- function(size,overlap,uniquePct=75) {
u1Size <- as.integer(size*uniquePct/100);
u1SizePerKeyColumn <- as.integer(ceiling(u1Size^(1/3)));
keys1 <- expand.grid(stringsAsFactors=F,
idCharacter=replicate(u1SizePerKeyColumn,paste(collapse='',sample(letters,sample(4:12,1L),T))),
idInteger=sample(u1SizePerKeyColumn),
idLogical=sample(c(F,T),u1SizePerKeyColumn,T)
)[seq_len(u1Size),];
keys1 <- rbind(keys1,keys1[sample(nrow(keys1),size-u1Size,T),])[sample(size),];
com <- as.integer(size*overlap);
uni <- size-com;
keys2 <- data.frame(stringsAsFactors=F,
idCharacter=replicate(uni,paste(collapse='',sample(letters,sample(4:12,1L),T))),
idInteger=u1SizePerKeyColumn+sample(uni),
idLogical=sample(c(F,T),uni,T)
);
keys2 <- rbind(keys2,keys1[sample(nrow(keys1),com,T),])[sample(size),];
keyNames <- c('idCharacter','idInteger','idLogical');
argSpecs <- list(
default=list(copySpec=1:2,args=list(
df1 <- cbind(stringsAsFactors=F,keys1,y1=sample(c(F,T),size,T),y2=sample(size),y3=rnorm(size),y4=replicate(size,paste(collapse='',sample(letters,sample(4:12,1L),T)))),
df2 <- cbind(stringsAsFactors=F,keys2,y5=sample(c(F,T),size,T),y6=sample(size),y7=rnorm(size),y8=replicate(size,paste(collapse='',sample(letters,sample(4:12,1L),T)))),
keyNames
)),
data.table.unkeyed=list(copySpec=1:2,args=list(
as.data.table(df1),
as.data.table(df2),
keyNames
)),
data.table.keyed=list(copySpec=1:2,args=list(
setkeyv(as.data.table(df1),keyNames),
setkeyv(as.data.table(df2),keyNames)
))
);
initSqldf();
sqldf(paste0('create index df1_key on df1(',paste(collapse=',',keyNames),');'));
sqldf(paste0('create index df2_key on df2(',paste(collapse=',',keyNames),');'));
argSpecs;
};
sizes <- c(1e1L,1e3L,1e5L);
overlaps <- c(0.99,0.5,0.01);
solTypes <- setdiff(getSolTypes(),'in.place');
system.time({ res <- testGrid(makeArgSpecs.assortedKey.optionalManyToMany,sizes,overlaps,solTypes); });
使用上述相同的绘图代码,得到的绘图如下:
titleFunc <- function(overlap) sprintf('R merge solutions: character/integer/logical key, 0..*:0..* cardinality, %d%% overlap',as.integer(overlap*100));
plotRes(res,titleFunc,F);