我有一组(2k - 4k)小字符串(3-6个字符),想要对它们进行聚类。由于使用了字符串,之前在如何进行聚类(特别是字符串聚类)中的答案告诉我,Levenshtein距离是用作字符串距离函数的好方法。而且,由于我事先不知道聚类的数量,层次聚类是正确的选择,而不是k-means。
尽管我理解了这个问题的抽象形式,但我不知道实际操作起来最简单的方法是什么。例如,MATLAB或R哪个更适合使用自定义函数(Levenshtein距离)实现分层聚类。 对于这两种软件,人们可以很容易地找到Levenshtein距离的实现。但聚类部分似乎更难。例如,在MATLAB中聚类文本计算所有字符串的距离数组,但我不知道如何使用距离数组来实际获得聚类结果。你们任何一位专家能否向我展示如何在MATLAB或R中使用自定义函数实现分层聚类呢?
尽管我理解了这个问题的抽象形式,但我不知道实际操作起来最简单的方法是什么。例如,MATLAB或R哪个更适合使用自定义函数(Levenshtein距离)实现分层聚类。 对于这两种软件,人们可以很容易地找到Levenshtein距离的实现。但聚类部分似乎更难。例如,在MATLAB中聚类文本计算所有字符串的距离数组,但我不知道如何使用距离数组来实际获得聚类结果。你们任何一位专家能否向我展示如何在MATLAB或R中使用自定义函数实现分层聚类呢?
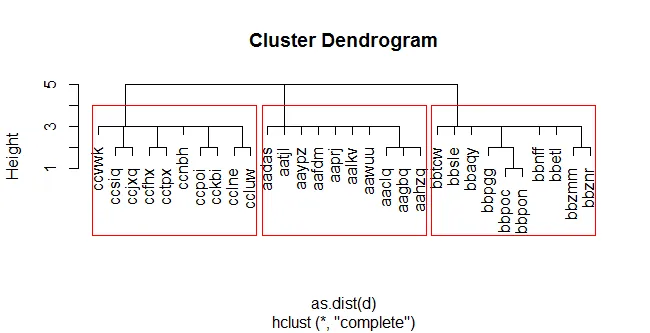
hclust)应该可以正常工作。另一方面,平均链接或Ward方法需要在每个步骤重新计算距离,因此它们将更加复杂。 - gung - Reinstate Monica